文章信息
- 孙晓洁, 高春辉, 黄巧云, 蔡鹏
- SUN Xiao-jie, GAO Chun-hui, HUANG Qiao-yun, CAI Peng
- 自然环境中的多物种生物膜: 研究方法及社群相互作用
- Multispecies Biofilms in Natural Environments: An Overview of Research Methods and Bacterial Social Interactions
- 农业资源与环境学报, 2017, 34(1): 6-14
- Journal of Agricultural Resources and Environment, 2017, 34(1): 6-14
- http://dx.doi.org/10.13254/j.jare.2016.0255
-
文章历史
- 收稿日期: 2016-10-21
生物膜(Biofilm)是微生物在固-液或气-液等界面聚集,并包被在自身分泌的胞外聚合物(Extracellu-lar polymeric substance,EPS)中而形成的微生物聚集体[1],其在自然和人体等环境中普遍存在。EPS能够维持生物膜的三维空间结构,为生物膜内部细胞间的交流和相互作用提供稳定的内环境[2]。在EPS 内往往包被多种微生物,包括不同的细菌、真菌和藻类等。目前研究比较多的生物膜体系主要是由细菌构成的单物种生物膜(Monospecies biofilms)和多物种生物膜(Multispecies biofilms)。多物种生物膜内含有多种细菌,它们彼此发生竞争、合作、信号传递等相互作用,从而赋予了生物膜独特的性质和功能[3]。相对于单物种生物膜和游离态细胞,多物种生物膜可以增加群落整体对抗生素的耐受性,避免原生动物的捕食,增加水平基因转移的频率等[4]。
土壤、水体、工程设备等环境中的各种生物和非生物因素影响着微生物间的相互作用,形成了具有不同功能特性的多物种生物膜,这些复杂的微生物群落又会对周围环境产生反馈调节,从而影响土壤质量和植物适应性等[5]。比如,植物根际分泌的多糖可以促进细菌生物膜的形成,而其中属于植物根际促生菌(Plant growth-promoting rhizobacteria,PGPR)的部分细菌又会促进植物生长[6]。多物种生物膜研究不仅能够帮助人们更好地认识微生物群落结构和生态功能,也可以为土壤环境中有机污染物降解、重金属转化、以及土传病害防治等提供新的思路。
在本文中,笔者首先论述了研究生物膜的不同方法,包括生物膜体系的构建以及分析和检测技术,特别介绍了这些方法在多物种生物膜研究中的一些应用。第二部分内容主要关注多物种生物膜中的相互作用包括种间和种内互作,并以合作和竞争作用为例,论述了多物种生物膜中互作的特点和意义。最后,指出了自然环境中多物种生物膜研究存在的问题和面临的挑战。
1 多物种生物膜研究方法生物膜的形成是一个十分复杂的过程,在实验室中构建生物膜体系会受到多种实验因素的影响。这些因素包括菌种类型及来源、接种量和接种时间、营养水平、外部环境的理化性质等。实验室条件下进行的多物种生物膜研究,通常包括实验菌株的选择,生物膜体系的构建以及检测分析技术。R覬der 等[7]指出在实验室中构建多物种生物膜群落时常用的3 种微生物选择标准分别为:(1)使用已鉴定的菌株集合,将来源不同的菌株放到一起进行培养;(2)利用分离自同一环境、并假定它们能够共存的菌株构建多物种生物膜;(3)直接使用没有经过任何培养步骤的复杂环境样品。本文主要关注生物膜体系的建立以及各种分析方法的比较(表 1)。通常,生物膜系统可以根据是否持续通入营养物质分为静态和动态两种。静态生物膜体系操作简单方便,而且可以检测多种不同条件下生物膜的形成情况。动态生物膜体系则能够更好地模拟自然环境,对于多物种生物膜的研究具有重要意义。生物膜的检测分析包括生物膜定量、结构观察以及功能分析。结晶紫染色是最常用的定量方法;利用扫描电子显微镜等显微技术可以实现对生物膜的直接观察[8],而各种分子生物学手段则常用于生物膜功能的分析。
静态生物膜体系常应用于形成生物膜的气-液或固-液界面,使细菌在界面上聚集形成生物膜。对于可以在气-液界面形成的生物膜,研究人员往往使用微孔板或者试管培养生物膜。近年来,改进自Galgary 生物膜设备[9]的Nunc-TSP 微孔板越来越多地用于培养细菌生物膜。Ren等[10]对普通96 孔板与Nunc-TSP微孔板进行了比较,结果发现Nunc-TSP 体系重复性更好。与微孔板相似,细菌也可以在试管内的气-液界面形成生物膜。关于Pseudomonas fluorescens 适应辐射的一些研究使用了含肉汤培养基的试管作为细菌生长的微环境[11]。这一体系简单且易于操作,不仅可以用于研究影响生物膜形成的因素,也可以用于研究生物膜内的物种多样性[12]。而对于可以在固体表面形成生物膜的细菌,则通常利用固体平板培养基观察菌落形态,判断生物膜形成能力。目前常用到的固体培养基包括MSgg 生物膜诱导培养基[13]、TSB 固体平板[14]、MB 平板[15]等。此外,固体平板上进行的群集实验(Swarming Assay,图 1(A)和泳动实验(Swimming As-say)也常用于不同细菌间相互作用的研究[16]。
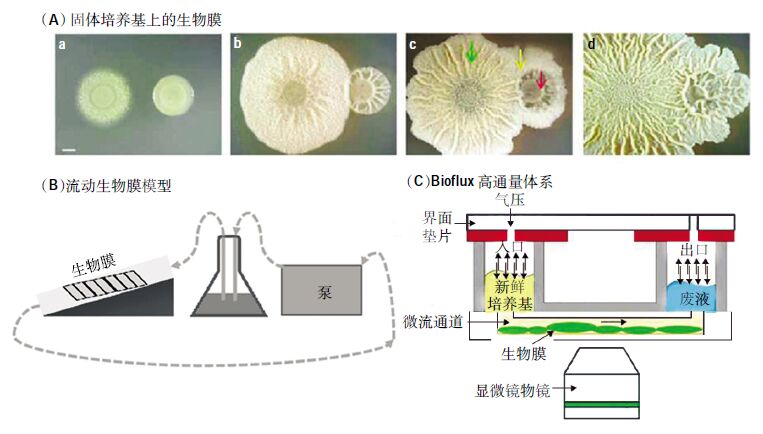
|
| A为MSgg生物膜诱导平板上的Bacillus subtilis 和Bacillus simplex生物膜,距离为0.8 cm[13]。(a)接触之前(第1 d),(b)接触(第2 d),(c)吞食(第3 d),(d)包围(第4 d)。箭头表示不同的互作区域:B. subtilis 区域(绿色箭头),界面(黄色箭头),B. simplex区域(红色箭头),比例为2 mm;B为流动生物膜体系[10]。琼脂覆盖的载玻片上通入无菌的土壤浸出液,流速为7.5 mL·min-1;C 为Bioflux高通量生物膜体系原理图[19]。气压传到入口顶部,使新鲜培养及进入含有生物膜的微孔道,并通过出口流出。生物膜可以直接用显微镜进行观察。 图 1 不同生物膜模型[10,13,19] Figure 1 Experimental models for studying biofilms[10,13,19] |
自然环境中的大部分生物膜是处于动态变化中的,它们可以从环境中得到持续地营养供给,因此在实验中需要尽量模拟自然动态体系。BurmØlle等[17]将琼脂覆盖的载玻片放入容器中,通过不断泵入无菌土壤浸出液作为细菌的营养来源,构建了一个闭合回路的生物膜流动模型(图 1(B))。这一模型模拟了细菌的自然生长环境,可以用于研究生物膜的演替过程及其影响因素。此外,由流动池组成的连续培养体系也可以用于构建生物膜模型。这一体系常与可以直接进行显微观察的培养室结合使用,但是往往是低通量的,不能同时进行多个样品的检测[7, 18]。而由微流通道组成的Bioflux 系统为生物膜的高通量分析奠定了基础,也为在微尺度下研究细菌间的相互作用提供了新的途径(图 1(C))[19]。利用流动池和Bioflux 体系对荧光标记菌株进行研究,使研究人员对多物种生物膜以及不同Pseudomonas aeruginosa菌株的分布和空间结构有了更深入的认识[20-21]。与静态方法不同的是,流动体系更加接近自然条件,能够实现对生物膜原位、持续地观测,适合研究生物膜形成和演替过程。
1.2 多物种生物膜的分析检测技术在多物种生物膜研究中,单菌种生物膜与多物种生物膜的生物量是生物膜分析的基本参数,其可以反映细菌成膜能力,也常作为判断相互作用类型的标准。对于微孔板中形成的生物膜,常使用结晶紫对其染色,然后检测590 nm处的吸光值,并将此作为生物膜的生物量参考值。这一方法操作简单,但是不能区分活细胞和死细胞,只是对生物膜生物量进行粗略定量。对于多物种生物膜中各物种的定量研究,过去一般将生物膜分离出来,进行平板计数,但是这一方法不适于复杂生物膜群落的研究,特别是天然生物膜群落中可能存在大量不可培养细菌,使用平板计数的方法往往会忽略掉这部分细菌的存在。通过定量PCR技术(quantitative Real-Time PCR,qPCR),利用特异引物对起始模板进行定量则可以完整地反映生物膜中各物种的含量。qPCR 技术已在牙菌斑生物膜[22]、水生生物膜[23]等生物膜研究中得到应用。在土壤多物种生物膜研究中,研究人员利用荧光染料SYBR Green I和特异性引物进行qPCR,得到了一个四菌种混合生物膜各物种的相对比例,发现了多物种生物膜形成过程中的关键菌种[10]。细菌的成膜能力除了用上述定量方法进行判断以外,固体培养基上形成的细菌菌落大小或者流动模型中生物膜的形态等也可以反映细菌的成膜能力,并且也能反映生物膜的结构。某些染料如刚果红可以与EPS 中的纤维素以及细菌菌毛等结合,将其加入到固体培养基中作为细菌运动的指示剂,可以清晰地反映生物膜的形成情况[24]。
生物膜作为细菌一种区别于游离态细胞的生命形式,其特定的空间结构也是研究人员关注的热点。固体培养基上细菌菌落是生物膜外部结构的直观反映,如B. subtilis 生物膜的褶皱[25]、Myxococcus xanthus的胞外多糖微孔道[26]等。要得到生物膜内部的空间构造,还需要更精细的可视化技术如扫描电子显微镜等[7, 27]。此外,通过荧光标记基因将特定菌种进行标记,利用荧光显微镜或者共聚焦激光扫描显微镜(Confocal laser scanning microscopy,CLSM)也可以清晰地观察到细菌生物膜的空间结构。CLSM 常与流动池相结合用于多物种生物膜研究。在实验中将载玻片放进流动池内,不断通入营养物质,利用CLSM 可以对载玻片上的生物膜进行观察[28]。此方法不会对生物膜造成破坏,而且可以实现对生物膜的连续监测,因此有助于剖析多物种生物膜的复杂空间结构及其时空演替过程。但是,很多微生物都不含报告基因,或者不适合进行荧光标记,所以仍需要对新的标记技术进行不断探索。
荧光原位杂交技术(Fluorescence in situ hybridiza-tion,FISH)则可以在原位得到样品的空间分布情况。Almstrand 等[29]在多物种生物膜荧光原位杂交和数据图像处理方面设计和应用了很多新方法。研究人员用FISH 技术在生物膜中检测到了多个种群,阐述了不同种群的层化现象。FISH 技术不依赖于细菌培养技术,可以对微生物群落的组成和动力学进行原位分析。而此技术的变体酶联免疫荧光原位杂交(Catalyzed reporter deposition FISH,CARD-FISH)可以得到更强、更稳定的荧光信号,常用于土壤等环境中不同微生物的检测[30];也可以与CLSM 结合,观察生物膜的种群空间分布[31]。
在研究多物种生物膜群落的多样性和演替过程时,往往需要对各组成细菌进行鉴定。DNA 抽提并结合变性梯度凝胶电泳(Denaturing gradient gel elec-trophoresis,DGGE)和16S rRNA 基因测序技术,可以得到细菌多样性,而不同时间段的分析结果可以反映生物膜的细菌群落演替过程[17]。但是DGGE 技术随机性较大,很难检测到低丰度菌种,甚至某些高丰度菌种也会出现丢失,而这些菌种有可能在生物膜形成中发挥重要作用。高通量测序能够较为全面和准确地反映微生物的群落结构,也可以在整体微生物群落水平分析物种遗传多样性,并能较为客观地反映其中低丰度的重要功能微生物,在微生物群落研究中已经有了较多应用。
与微生物的纯培养相对应,很多分子生物学方法可以用于检测生物膜相关基因的表达调控,特别是关于B. subtilis、P. aeruginosa、Vibrio cholerae 等单菌种生物膜的相关研究[32-34]。单核苷酸多态性(Single nu-cleotide polymorphism,SNP)等分子手段也逐渐用于检测生物膜中存在的基因突变、氨基酸移码等[35-36]。基因芯片技术具有高通量、大规模、平行性地分析基因表达的能力,可以用于筛选生物膜中特异表达的基因。而蛋白组学与高效质谱分析技术相结合,则能够用于比较游离态细胞与生物膜细胞在蛋白质组成和数量上的差异[37]。各种比较组学技术的发展和应用对于深入了解微生物群落生态学具有重要意义[38-39]。它们与网络分析方法一起,可以用于确定生物膜形成的关键菌种,构建模式生物膜体系[7, 40],在研究生物膜特别是多物种生物膜的形成和演替过程中具有广泛的应用前景。
2 多物种生物膜中的相互作用Parsek 等[41]提出了“微生物社会学(Sociomicrobi-ology)”一词,特别强调了微生物群落及其内部各物种间各种相互作用和信息交流的重要性。生物膜也是微生物群落的一种存在状态。自然界中的生物膜群落大都含有多种微生物,它们彼此进行信息传递,发生相互作用,最终形成稳定的生态体系。多物种生物膜中的相互作用包括细菌间的合作和竞争,信号分子的传递以及水平基因转移等。生物膜群落内部包含高度的物种多样性和异质性,其结构也会受到各种生物、化学和物理等因素的影响[42]。物种的多样性导致生物膜中相互作用类型的复杂性,而其中各种物质的异质性则会影响不同信号间的传递以及水平基因转移频率。
在本文中,笔者将针对竞争和合作作用进行详细论述。竞争和合作作为生物膜中的主要相互作用类型,可以发生在种内,也可以发生在不同菌种之间。在研究生物膜时,多物种生物膜生物量与各物种形成的单菌种生物膜生物量的比较,往往被用作判断各组分互作类型的依据。一般认为,微生物间的合作是对发生相互作用的受体菌有利的行为,而竞争作用则是对受体菌不利的行为。Madsen 等[43]也提出了一种更精确的定义方式:多物种生物膜的量高于各单菌种生物膜生物量之和时,为协同效应;而比成膜能力最差的细菌所形成的生物膜的量还低时,则认为它们之间为拮抗互作(图 2)。可以借鉴此定义方法,对多物种生物膜种间互作类型进行判断。
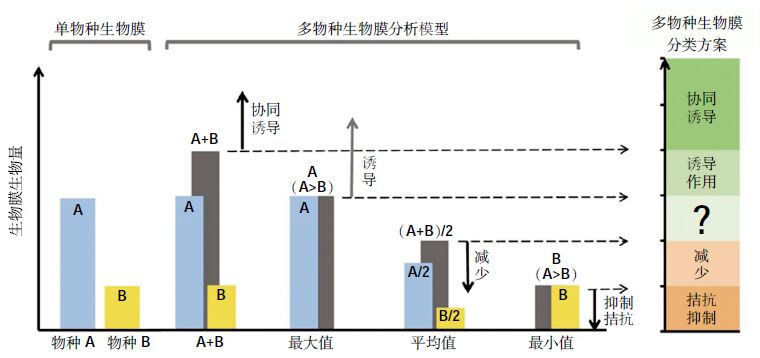
|
| 蓝色:A菌成膜能力;黄色:B菌成膜能力;黑色:用于定义细菌A和细菌B 共培养时的成膜能力。诱导作用:共培养时的成膜能力大于单独培养时的最大成膜能力。生物膜减少:共培养时产生的生物膜小于单独培养时的均值。因为某些相互作用不能直接观察,所以中间未定义的区域保证了此模型的严谨性。此分类方法可以用于任意种类细菌形成的多物种生物膜 图 2 以单菌种生物膜形成情况为基础的多物种生物膜相互作用分类方法[43] Figure 2 Classification scheme used for the assessment of biofilm formation in co-cultures based on monoculture biofilm formation[43] |
生物膜中的细菌彼此接近,关系密切,为细菌间的互作提供了前提。生物膜内的细菌可以利用其他细菌的代谢产物或者一起共同抵御不利环境。在一个由Pseudomonas putida SB5 和Chryseobacterium sp. SB9构成的双菌种生物膜中,其生物量相对于单菌种生物膜生物量显著增加,这说明这两个菌株之间存在协同互作;P. putida SB5能够促进Chryseobacteriumsp. SB9生物膜的形成。此外,研究人员还发现,这两个细菌在与其他菌株混合培养时不会出现生物膜量增加的现象,这表明它们之间的互作具有菌株特异性[17]。生物膜中存在的协同效应也能促进整个群落对表面活性剂[28]、抗生素[44]、农药[45]等外界环境压力的耐受性或降解能力。
除此之外,也有很多研究观察到多物种生物膜的协同作用可以促进整体生物膜的形成[46-47]。研究人员从废水处理系统中分离到13株细菌,分别进行两两混合培养,发现多物种生物膜具有不同于单菌种的独特性质,不同细菌间存在协同效应[48]。Ren 等[49]利用四菌种混合培养的方法,研究了土壤中7 株不同的细菌在生物膜形成过程中的作用。结果发现有63%的四菌种混合培养物的生物膜量较单菌种生物膜出现了增加,这表明协同作用在多物种生物膜形成过程中普遍存在。由Stenotrophomonas rhizophila,Xan 原thomonas retroflexus,Microbacterium oxydans 和Paeni-bacillus amylolyticus 组成的混合培养物协同作用非常显著。菌株特异性qPCR 结果表明,X. retroflexus 在混合培养物中含量最多(大于生物膜总细胞数的97%),其余3 个菌株虽然相对丰度较低,但它们是多物种生物膜中细菌协同作用所必需的。
生物膜是不同细菌进行代谢产物交换的场所,其中某些细菌将其他细菌的代谢产物为己所用,如作为电子受体或供体等[50-51]。所以,目前普遍认为代谢相关性是多物种生物膜内不同细菌间彼此合作的原因之一。硝化过程就是生物膜中不同微生物彼此进行代谢合作的一个典型例子。在这一过程中,氨氧化细菌将铵盐转化为亚硝酸盐,随后又被亚硝酸盐氧化细菌氧化[52]。此外,产脲酶的Nitrospira moscoviensis 会合成氨供不产脲酶的氨氧化细菌氧化所用[8, 52]。生物膜中的氨氧化细菌和亚硝酸盐氧化细菌利用彼此的代谢产物,从而实现代谢合作。
2.2 生物膜的种间和种内竞争竞争作用也是多物种生物膜中常见的互作方式。生物膜中的各种细菌可能彼此竞争营养物质、溶解氧以及空间等,并抑制其他细菌的生长繁殖。最常见的例子就是废水处理系统中各种微生物群落的演替过程。在下水道内壁由硫酸盐还原细菌(Sulfate-reducing bacteria,SRB)与产甲烷古菌(Methanogenic ar-chaea,MA)构成的厌氧生物膜中,SRB 和MA 彼此竞争有机质、醋酸盐和氢等基质。在生物膜形成早期,生物膜中的主要微生物是SRB,但是在生物膜成熟后,CH4的排放速率显著增加,MA 成为了生物膜中的主要微生物[53]。此外,微生物间的竞争作用也包括分泌毒性物质如抗生素、细菌素、生物表面活性剂等直接杀死或抑制其他细菌的生长。比如,P. aeruginosa等致病菌的VI型分泌系统(Type VI secretion system,T6SS)可以将毒力效应蛋白转入不具T6SS 的邻近细胞内,从而抑制甚至杀死周围细菌[54-55]。一些属于Bacillus和Pseudomonas 的生防细菌(Biocontrol bacteria)也可以分泌2,4-二乙酰基间苯三酚(2,4-diacetylphloroglu-cinol,2,4-DAPG)等具有抗菌活性的物质,从而抑制植物病原菌的生长[56]。
事实上,种间和种内微生物均存在竞争关系。据报道,固体培养基上的B. subtilis 可以吞食其他竞争性的B.simplex菌落(图 1. A)[13]。一方面是因为B. subtilis可以分泌不利于B. simplex 生长的表面活性素和自食毒素。而且,在生物膜形成和种间互作过程中,B.subtilis 生物膜中的一个亚群丢失了携带有抑制这两种毒性物质合成的原生质粒pBS32,这也增加了其对其他竞争性芽孢杆菌的毒性。Oliveira 等[20]指出,种内竞争作用也在多物种生物膜形成和维持群落功能上发挥重要作用。在一系列P. aeruginosa天然菌株的不同组合中,不仅多物种生物膜的生物量出现了增加,而且混合培养的不同菌株间产生了更强的竞争作用。一株菌在生物膜中占优势,且会在很大程度上排斥另一株细菌,这表明生物膜的增加是细菌对竞争作用的响应。
多样性是不同生态体系的基本特征。物种多样性是影响群落功能和稳定性的关键因素,而基因型的多样性在提高种群性能方面发挥重要作用。在一个群落中,这两者之间往往存在相互作用,而且会影响整个群落的性质和功能。多物种生物膜作为一个由不同微生物构成的群落,其中的种间多样性会使种内多样性减少并在功能上取代种内多样性[57]。研究人员构建了由P. aeruginosa PAO1,Pseudomonas protegens Pf-5,Klebsiella pneumoniae KP-1三种细菌组成的多物种生物膜群落,发现种间多样性在多物种生物膜群落中占主要地位,而形态学变种的出现则会降低群落对十二烷基硫酸钠的耐受性,这表明种间多样性可在功能上代替种内多样性。自然环境中的多物种生物膜群落中往往存在高度的种间多样性,而形态学变种可以忽略不计。但是,一些种间多样性较低的特定环境如囊性纤维化病人肺部等,仍存在形态学变种,且发挥重要作用[58]。所以,研究多物种生物膜时应该根据不同环境给予种间和种内多样性特定关注,这样可以简化研究模型,有利于不同互作机制的阐释。
2.3 竞争和合作,谁才是主角?目前很多研究都强调了生物膜内各菌种间协同合作的重要性及普遍性[59],但是也有部分研究发现了与此相反的结果。Foster 等[60]指出生物膜中的种间互作主要是竞争作用。研究人员将来源于同一水生环境的菌株两两组合时,发现它们的互作大多都是负效应;即使是两种以上菌种的混合也没有产生正向效应。这说明生物膜中各种细菌相互适应的结果通常是产生竞争性表型,而非合作表型。此外,Oliveira 等[20]发现在抗生素胁迫下生物膜的增加也表现为细菌对竞争作用的响应。
必须要强调的一点是,生物膜内的细菌彼此合作,又相互竞争。当组成多物种生物膜的细菌均来源于同一环境时,多物种生物膜中的各菌种间更容易进行合作;但是,如果它们来源于不同环境,其中的合作则非常弱[4, 43]。另一方面,生物膜中各物种的亲缘关系也会影响其相互作用。如果互作细菌在决定某一群落性质的基因座上具有相同的基因型,则更有利于彼此合作;若这些细菌在这一基因座上属于不同基因型,则常会产生竞争作用[61]。此外,生物膜中不同细胞的空间组织情况也会对合作和竞争产生影响[54]。空间受限制时,生物膜内部呈现为不同细胞系的隔离状态,邻近细胞几乎完全来源于同一克隆,所以更容易进行合作;而生物膜内部细胞高度混合时,互作往往以拮抗为主,当然也可能存在不同菌株间的共生作用。
Zelezniak等[62]指出,代谢合作是不同菌种能够在群落中共存的主要原因。在一个由B. subtilis 构成的生物膜中,研究人员发现生物膜周围和内部的细胞之间具有代谢相关性。位于生物膜周围的细胞一方面能够保护内部细胞不受外界环境干扰,也会通过消耗养分来抑制内部细胞的生长。目前普遍认为这种代谢相关性是平衡竞争与合作的关键[63]。代谢的耦联也体现为多物种生物膜中微生物基因表达的协同变化[64],但是这种代谢相关性在多物种生物膜中是否普遍存在仍有待进一步研究。
3 结论与展望无论是有害生物膜的抑制,还是有益生物膜的利用,都需要高度关注多物种生物膜中微生物间的相互作用。然而,目前该领域研究仍面临巨大挑战。一方面缺少合适的尺度范围和标准方法,无法在实验室实现环境中生物膜复杂微环境的重现,进而对生物膜的原位研究较少,研究方法和相关技术仍有待完善。另一方面,虽然实验室中基于可培养技术的生物膜体系的构建使研究简单化,但是往往会忽略掉不可培养细菌在多物种生物膜中发挥的重要作用。而且,生物膜内的微生物彼此合作,又相互竞争,它们如何在生物膜内达到平衡以及这一平衡如何影响生物膜的功能特征也需要进行深入探索。此外,目前虽然对单菌种生物膜形成过程已经有了一定的认识,发现了一些生物膜调控基因,但多物种生物膜形成的分子机制仍不清楚。
未来研究人员应更关注自然环境中的复杂生物膜群落,模拟自然环境建立流动态生物膜模式体系,或者在原位揭示多物种生物膜中各菌种的演替及其生态学特征和效应。生物膜内各物种之间相互作用协调机制的阐释还需要一个从量变到质变的过程。随着研究的不断增多和深入,可以在大量实验数据的基础上阐释多物种生物膜普遍存在的互作类型。利用高通量测序可以较为全面和准确地反映微生物的群落结构,对于研究多物种生物膜的演替和种间互作机制具有重要意义。在分子生物学方面,运用多种宏组学方法可以研究细胞不同阶段的基因表达差异,分析其调控过程,从而从根本上解释生物膜的形成和演替机制。
| [1] | Hall-Stoodley L, Costerton J W, Stoodley P. Bacterial biofilms: From the natural environment to infectious diseases[J]. Nature Reviews Microbiology , 2004, 2 (2) : 95–108. DOI:10.1038/nrmicro821 |
| [2] | Flemming H-C, Wingender J. The biofilm matrix[J]. Nature Reviews Microbiology , 2010, 8 : 623–633. |
| [3] | Rendueles O, Ghigo J-M. Multi-species biofilms: How to avoid unfriendly neighbors[J]. FEMS Microbiology Reviews , 2012, 36 (5) : 972–989. DOI:10.1111/j.1574-6976.2012.00328.x |
| [4] | Burmølle M, Ren D, Bjarnsholt T, et al. Interactions in multispecies biofilms: Do they actually matter?[J]. Trends in Microbiology , 2014, 22 (2) : 84–91. DOI:10.1016/j.tim.2013.12.004 |
| [5] | Barea J-M. Microbial co-operation in the rhizosphere[J]. Journal of Experimental Botany , 2005, 56 (417) : 1761–1778. DOI:10.1093/jxb/eri197 |
| [6] | Beauregard P B, Chai Y, Vlamakis H, et al. Bacillus subtilis biofilm induction by plant polysaccharides[J]. Proceedings of the National Academy of Sciences , 2013, 110 (17) : E1621–E1630. DOI:10.1073/pnas.1218984110 |
| [7] | Røder H L, Sørensen S J, Burmølle M. Studying bacterial multispecies biofilms: Where to start?[J]. Trends in Microbiology , 2016, 24 (6) : 503–513. DOI:10.1016/j.tim.2016.02.019 |
| [8] | Hu H, Johani K, Gosbell I B, et al. Intensive care unit environmental surfaces are contaminated by multidrug-resistant bacteria in biofilms: Combined results of conventional culture, pyrosequencing, scanning electron microscopy, and confocal laser microscopy[J]. Journal of Hospital Infection , 2015, 91 (1) : 35–44. DOI:10.1016/j.jhin.2015.05.016 |
| [9] | Ceri H, Olson ME, Stremick C, et al. The calgary biofilm device: New technology for rapid determination of antibiotic susceptibilities of bacterial biofilms[J]. Journal of Clinical Microbiology , 1999, 37 (6) : 1771–1776. |
| [10] | Ren D, Madsen J S, de la Cruz-Perera C I, et al. High-throughput screening of multispecies biofilm formation and quantitative PCR -based assessment of individual species proportions, useful for exploring interspecific bacterial interactions[J]. Microbial Ecology , 2014, 68 (1) : 146–154. DOI:10.1007/s00248-013-0315-z |
| [11] | Spiers A J. A mechanistic explanation linking adaptive mutation, niche change, and fitness advantage for the wrinkly spreader[J]. International Journal of Evolutionary Biology , 2014, 2014 : 1–10. |
| [12] | Steenackers H P, Parijs I, Foster K R, et al. Experimental evolution in biofilmpopulations[J]. FEMS Microbiology Reviews , 2016, 40 (3) : 373–397. DOI:10.1093/femsre/fuw002 |
| [13] | Rosenberg G, Steinberg N, Oppenheimer-Shaanan Y, et al. Not so simple, not so subtle: The interspecies competition between Bacillus simplex and Bacillus subtilis and its impact on the evolution of biofilms[J]. Biofilms and Microbiomes , 2016, 2 (3) : 753–762. |
| [14] | Koch G, Yepes A, Förstner KU, et al. Evolution of resistance to a lastresort antibiotic in Staphylococcus aureus via bacterial competition[J]. Cell , 2014, 158 (5) : 1060–1071. DOI:10.1016/j.cell.2014.06.046 |
| [15] | Michæl V, Frank O, Bartling P, et al. Biofilm plasmids with a rhamnose operon are widely distributed determinants of the "swim-or -stick" lifestyle in roseobacters[J]. The ISME Journal , 2016, 10 : 2498–2513. DOI:10.1038/ismej.2016.30 |
| [16] | Stefanic P, Kraigher B, Lyons N A, et al. Kin discrimination between sympatricBacillus subtilis isolates[J]. Proceedings of the National Academy of Sciences , 2015, 112 (45) : 14042–14047. DOI:10.1073/pnas.1512671112 |
| [17] | Burmølle M, Hansen L H, Sørensen S J. Establishment and early succession of a multispecies biofilm cmposed of soil bacteria[J]. Microbial Ecology , 2007, 54 (2) : 352–362. DOI:10.1007/s00248-007-9222-5 |
| [18] | Pamp S J, Sternberg C, Tolker-Nielsen T. Insight into the microbial multicellular lifestyle via flow-cell technology and confocal microscopy[J]. Cytometry Part A , 2009, 75A (2) : 90–103. DOI:10.1002/cyto.a.v75a:2 |
| [19] | Benoit M R, Conant C G, Ionescu-Zanetti C, et al. New device for high-throughput viability screening of flow biofilms[J]. Applied and Environmental Microbiology , 2010, 76 (13) : 4136–4142. DOI:10.1128/AEM.03065-09 |
| [20] | Oliveira N M, Martinez-Garcia E, Xavier J, et al. Biofilm formation as a response to ecological competition[J]. PLOS Biology , 2015, 13 (7) : e1002191. DOI:10.1371/journal.pbio.1002191 |
| [21] | Hansen S K, Rainey P B, Haagensen J A J, et al. Evolution of species interactions in a biofilmcommunity[J]. Nature , 2007, 445 (7127) : 533–536. DOI:10.1038/nature05514 |
| [22] | Kirakodu S S, Govindaswami M, NovakM J, et al. Optimizing qPCR for the quantification of periodontal pathogens in a complex plaque biofilm[J]. The Open Dentistry Journal , 2008, 2 (1) : 49–55. DOI:10.2174/1874210600802010049 |
| [23] | Beumer A, King D, Donohue M, et al. Detection of Mycobacterium avium subsp. paratuberculosis in drinking water and biofilms by quantitative PCR[J]. Applied and Environmental Microbiology , 2010, 76 (2) : 7367–7370. |
| [24] | Wu Y, Ding Y, Cohen Y, et al. Elevated level of the second messenger c-di-GMP in Comamonas testosteroni enhances biofilm formation and biofilm-based biodegradation of 3-chloroaniline[J]. Applied Microbiology and Biotechnology , 2015, 99 (4) : 1967–1976. DOI:10.1007/s00253-014-6107-7 |
| [25] | Vlamakis H, Chai Y, Beauregard P, et al. Sticking together: Building a biofilmthe Bacillus subtilis way[J]. Nature Reviews Microbiology , 2013, 11 (3) : 157–168. DOI:10.1038/nrmicro2960 |
| [26] | Berleman J E, Zemla M, Remis J P, et al. Exopolysaccharide microchannels direct bacterial motility and organize multicellular behavior[J]. The ISME Journal , 2016, 10 : 2620–2632. DOI:10.1038/ismej.2016.60 |
| [27] | Eick S, Markauskaite G, Nietzsche S, et al. Effect of photoactivated disinfection with a light -emitting diode on bacterial species and biofilms associated with periodontitis and peri-implantitis[J]. Photodiagnosis and Photodynamic Therapy , 2013, 10 (2) : 156–167. DOI:10.1016/j.pdpdt.2012.12.001 |
| [28] | Lee KWK, Periasamy S, Mukherjee M, et al. Biofilm development and enhanced stress resistance of a model, mixed -species community biofilm[J]. The ISME Journal , 2014, 8 (4) : 894–907. DOI:10.1038/ismej.2013.194 |
| [29] | Almstrand R, Daims H, Persson F, et al. New methods for analysis of spatial distribution and coaggregation of microbial populations in complex biofilms[J]. Applied and Environmental Microbiology , 2013, 79 (19) : 5978–5987. DOI:10.1128/AEM.01727-13 |
| [30] | Amann R, Fuchs B M. Single-cell identification in microbial communities by improved fluorescence in situ hybridization techniques[J]. Nature Reviews Microbiology , 2008, 6 (5) : 339–348. DOI:10.1038/nrmicro1888 |
| [31] | Bulgarelli D, Rott M, Schlæppi K, et al. Revealing structure and assembly cues for Arabidopsis root-inhabiting bacterial microbiota[J]. Nature , 2012, 488 (7409) : 91–95. DOI:10.1038/nature11336 |
| [32] | Mielich-Süss B, Lopez D. Molecular mechanisms involved in Bacillus subtilis biofilm formation[J]. Environmental Microbiology , 2015, 17 (3) : 555–565. DOI:10.1111/emi.2015.17.issue-3 |
| [33] | Burrows L L. Pseudomonas æruginosa twitching motility: Type IV Pili in action[J]. Annual Review of Microbiology , 2012, 66 (1) : 493–520. DOI:10.1146/annurev-micro-092611-150055 |
| [34] | Teschler J K, Zamorano-Sánchez D, Utada A S, et al. Living in the matrix: Assembly and control of Vibrio choleræ biofilms[J]. Nature Reviews Microbiology , 2015, 13 (5) : 255–268. DOI:10.1038/nrmicro3433 |
| [35] | Selezska K, Kazmierczak M, Müsken M, et al. Pseudomonas æruginosa population structure revisited under environmental focus: Impact of water quality and phage pressure: Environmental P. æruginosa population structure[J]. Environmental Microbiology , 2012, 14 (8) : 1952–1967. DOI:10.1111/j.1462-2920.2012.02719.x |
| [36] | Gill S R, Fouts D E, Archer G L, et al. Insights on evolution of virulence and resistance from the complete genome analysis of an early methicillin-resistant Staphylococcus aureus strain and a biofilm-producing methicillin-resistant Staphylococcus epidermidis strain[J]. Journal of Bacteriology , 2005, 187 (7) : 2426–2438. DOI:10.1128/JB.187.7.2426-2438.2005 |
| [37] | Park A J, Murphy K, Krieger J R, et al. A temporal examination of the planktonic and biofilm proteome of whole cell Pseudomonas æruginosa PAO1 using quantitative mass spectrometry[J]. Molecular & Cellular Proteomics , 2014, 13 (4) : 1095–1105. |
| [38] | Morris R M, Nunn B L, Frazar C, et al. Comparative metaproteomics reveals ocean-scale shifts in microbial nutrient utilization and energy transduction[J]. The ISME Journal , 2010, 4 (5) : 673–685. DOI:10.1038/ismej.2010.4 |
| [39] | Roume H, Heintz-Buschart A, Muller E E L, et al. Comparative integrated omics: Identification of key functionalities in microbial community-wide metabolic networks[J]. Biofilms and Microbiomes , 2015, 1 : 15007. DOI:10.1038/npjbiofilms.2015.7 |
| [40] | Barberán A, Bates S T, Casamayor E O, et al. Using network analysis to explore co-occurrence patterns in soil microbial communities[J]. The ISME Journal , 2012, 6 (2) : 343–351. DOI:10.1038/ismej.2011.119 |
| [41] | Parsek M R, Greenberg E P. Sociomicrobiology: The connections between quorum sensing and biofilms[J]. Trends in Microbiology , 2005, 13 (1) : 27–33. DOI:10.1016/j.tim.2004.11.007 |
| [42] | Ainslie E F Little, Robinson C J, Peterson S B, et al. Rules of engagement: Interspecies interactions that regulate microbial communities[J]. Annual Review of Microbiology , 2008, 62 (1) : 375–401. DOI:10.1146/annurev.micro.030608.101423 |
| [43] | Madsen J S, Røder H L, Russel J, et al. Coexistence facilitates interspecific biofilm formation in complex microbial communities: Coexistence promotes biofilm formation[J]. Environmental Microbiology , 2016, 18 (8) : 2565–2574. DOI:10.1111/1462-2920.13335 |
| [44] | Olsen I. Biofilm-specific antibiotic tolerance and resistance[J]. European Journal of Clinical Microbiology & Infectious Diseases , 2015, 34 (5) : 877–886. |
| [45] | Breugelmans P, Barken K B, Tolker-Nielsen T, et al. Architecture and spatial organization in a triple-species bacterial biofilm synergistically degrading the phenylurea herbicide linuron: Linuron-degrading bacterial biofilms[J]. FEMS Microbiology Ecology , 2008, 64 (2) : 271–282. DOI:10.1111/fem.2008.64.issue-2 |
| [46] | Burmolle M, Webb J S, Rao D, et al. Enhanced biofilm formation and increased resistance to antimicrobial agents and bacterial invasion are caused by synergistic interactions in multispecies biofilms[J]. Applied and Environmental Microbiology , 2006, 72 (6) : 3916–3923. DOI:10.1128/AEM.03022-05 |
| [47] | Røder H L, Raghupathi P K, Herschend J, et al. Interspecies interactions result in enhanced biofilm formation by co-cultures of bacteria isolated from a food processing environment[J]. Food Microbiology , 2015, 51 : 18–24. DOI:10.1016/j.fm.2015.04.008 |
| [48] | Andersson S, Kuttuva Rajarao G, Land C J, et al. Biofilm formation and interactions of bacterial strains found in wastewater treatment systems: Biofilm formation and interactions of bacterial strains[J]. FEMS Microbiology Letters , 2008, 283 (1) : 83–90. DOI:10.1111/fml.2008.283.issue-1 |
| [49] | Ren D, Madsen J S, Sørensen S J, et al. High prevalence of biofilmsynergy among bacterial soil isolates in cocultures indicates bacterial interspecific cooperation[J]. The ISME Journal , 2015, 9 (1) : 81–89. DOI:10.1038/ismej.2014.96 |
| [50] | West S A, Griffin A S, Gardner A, et al. Social evolution theory for microorganisms[J]. Nature Reviews Microbiology , 2006, 4 (8) : 597–607. DOI:10.1038/nrmicro1461 |
| [51] | McGlynn S E, Chadwick G L, Kempes C P, et al. Single cell activity reveals direct electron transfer in methanotrophic consortia[J]. Nature , 2015, 526 (7574) : 531–535. DOI:10.1038/nature15512 |
| [52] | Flemming H-C, Wingender J, Szewzyk U, et al. Biofilms: An emergent form of bacterial life[J]. Nature Reviews Microbiology , 2016, 14 (9) : 563–575. DOI:10.1038/nrmicro.2016.94 |
| [53] | Auguet O, Pijuan M, Batista J, et al. Changes in microbial biofilm communities during colonization of sewer systems[J]. Applied and Environmental Microbiology , 2015, 81 (20) : 7271–7280. DOI:10.1128/AEM.01538-15 |
| [54] | Nadell C D, Drescher K, Foster K R. Spatial structure, cooperation and competition in biofilms[J]. Nature Reviews Microbiology , 2016, 14 (9) : 589–600. DOI:10.1038/nrmicro.2016.84 |
| [55] | Basler M, Ho B T, Mekalanos J J. Tit-for-tat: Type VI secretion system counterattack during bacterial cell-cell interactions[J]. Cell , 2013, 152 (4) : 884–894. DOI:10.1016/j.cell.2013.01.042 |
| [56] | Raaijmakers J M, Mazzola M. 1. Diversity and natural functions of antibiotics produced by beneficial and plant pathogenic bacteria[J]. Annual Review of Phytopathology , 2012, 50 (1) : 403–424. DOI:10.1146/annurev-phyto-081211-172908 |
| [57] | Kelvin Lee K W, Hoong Yam J K, Mukherjee M, et al. Interspecific diversity reduces and functionally substitutes for intraspecific variation in biofilmcommunities[J]. The ISME Journal , 2016, 10 (4) : 846–857. DOI:10.1038/ismej.2015.159 |
| [58] | Woo J K K, Webb J S, Kirov S M, et al. Biofilm dispersal cells of a cystic fibrosis Pseudomonas æruginosa isolate exhibit variability in functional traits likely to contribute to persistent infection[J]. FEMS Immunology&Medical Microbiology , 2012, 66 (2) : 251–264. |
| [59] | Elias S, Banin E. Multi-species biofilms: living with friendly neighbors[J]. FEMS Microbiology Reviews , 2012, 36 (5) : 990–1004. DOI:10.1111/j.1574-6976.2012.00325.x |
| [60] | Foster K R, Bell T. Competition, not cooperation, dominates interactions among culturable microbial species[J]. Current Biology , 2012, 22 (19) : 1845–1850. DOI:10.1016/j.cub.2012.08.005 |
| [61] | Mitri S, Richard Foster K. The genotypic view of social interactions in microbial communities[J]. Annual Review of Genetics , 2013, 47 (1) : 247–273. DOI:10.1146/annurev-genet-111212-133307 |
| [62] | Zelezniak A, Andrejev S, Ponomarova O, et al. Metabolic dependencies drive species co-occurrence in diverse microbial communities[J]. Proceedings of the National Academy of Sciences , 2015, 112 (20) : 6449–6454. DOI:10.1073/pnas.1421834112 |
| [63] | Liu J, Prindle A, Humphries J, et al. Metabolic co-dependence gives rise to collective oscillations within biofilms[J]. Nature , 2015, 523 (7562) : 550–554. DOI:10.1038/nature14660 |
| [64] | Hansen L B S, Ren D, Burmølle M, et al. Distinct gene expression profile of Xanthomonas retroflexus engaged in synergistic multispecies biofilm formation[J]. The ISME Journal[Internet], 2016, Available from: http://www.nature.com/doifinder/10.1038/ismej.2016.107. |
 2017, Vol. 34
2017, Vol. 34





