2. 农业农村部华北耕地保育重点实验室, 北京 100193;
3. 自然资源部农用地质量与监控重点实验室, 北京 100035
2. Key Laboratory of Arable Land Conservation in North China, Ministry of Agriculture and Rural Affairs, Beijing 100193, China;
3. Key Laboratory of Land Quality, Ministry of Natural Resources, Beijing 100035, China
抗生素以其优异的性能被广泛用于人类和动物疾病治疗、促生长等方面[1],但是,人畜摄入的抗生素最终会以原型或代谢产物的形式通过排泄物进入到土壤和其他环境介质中[2-4],影响原有生态系统的结构与稳定性[5]。随着抗生素的大量生产、使用甚至滥用,抗生素和抗性基因污染已经成为一个全球性的环境健康问题[6]。自20世纪40年代起,Waksman等[7]率先开展了对土壤中抗生素的系统研究,随后各研究团队相继跟进,经过几十年的发展,在土壤中抗生素的发现与分离,抗生素及其抗性基因的种类、丰度分析,积累、转归等过程探索,以及生态风险评估与控制等方面取得丰硕成果。因此,总结土壤中抗生素研究的阶段性研究成果,梳理其整体脉络,分析研究热点与演进趋势,对全面了解并持续推进该领域的研究具有重要意义。
本研究基于Web of Science Core Collection论文数据库的文献源,借助文献计量学和知识图谱方法进行信息挖掘,立足于多元时空的动态视角,对土壤中抗生素研究的相关文献进行图谱分析,梳理国际上关于土壤中抗生素研究的现状与发展历程,归纳其研究核心与热点前沿方向,从而完整呈现土壤中抗生素研究的总体样貌,以期为研究者准确把握该领域的研究进展提供理论参考。
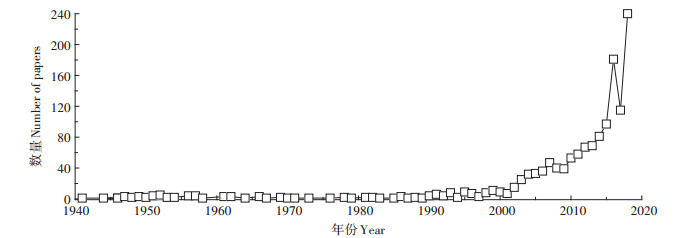
1 数据来源与方法 1.1 数据来源本研究以Web of Science为文献检索平台,选择“Web of Science Core Collection”数据库为样本框,检索策略设定为TS=(Antibiotic* and Soil),文献类型设定为“Article”,检索时间跨度为1900—2018年(检索时间为2019年6月3日),得到5 405个检索结果;对检索结果逐条筛选,剔除不相关的文章以及书评、卷首语等非学术性文章,并删除重复文献,最终得到1 355篇相关有效文献。将筛选后的文献以“摘要、全记录(包含引用的参考文献)”的格式下载保存为纯文本文件,作为分析数据样本。土壤中抗生素研究最早的相关文献始于1941年,而后相关的研究文献数量逐年增多(图 1);从文章的数量变化趋势来看,土壤中抗生素的研究仍处于较高增长期,充分说明土壤中抗生素的相关研究已成为环境领域的热点。
|
图 1 土壤中抗生素的研究发文量变化趋势 Figure 1 The number variation tendency of published articles of antibiotics in soil |
借助信息可视化软件CiteSpace进行分析,在软件中分别选择Author(作者)、Institution(机构)、Keyword(关键词)、Cited Reference(被引文献)等选项,节点强度默认Cosine(余弦函数)和Within slices(时间切片内),阈值选择TOP 50,网络裁剪功能区参数选择最小生成树(Minimum spanning tree,MST)进行图谱分析,得到土壤中抗生素研究领域的统计数据与共现图谱,分析其研究现状、热点与前沿。
在知识图谱中,density(网络密度)表示节点之间的联系强度;节点(字体)大小表示作者/机构/关键词的频次差异,节点越大,出现的频次越高;节点的圆圈层代表年轮,紫色的圆圈层为重点标注的中心性,年轮宽度可以指代中心性的大小,来反映作者/机构/关键词等的结构性和影响力[8];节点的颜色从紫色到淡黄色的变化代表研究的时间由远及近;节点连线表示共现频次,连线越粗合作越多。
2 结果与讨论 2.1 土壤中抗生素的研究路径演进研究主题的演进可以清楚地揭示研究领域的结构脉络及特点,鉴于关键词对文章主题的高度概括作用[9],借助CiteSpace软件的关键词分析工具所呈现的文章更新和相互影响情况,来揭示土壤中抗生素研究的演变过程。
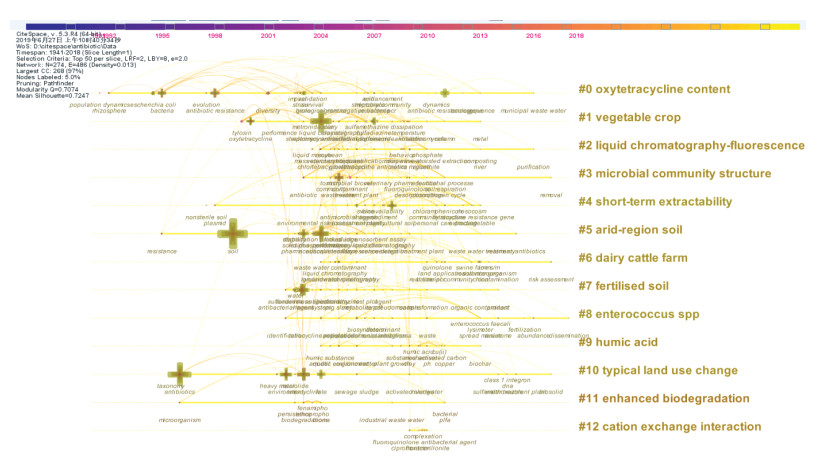
对软件分析得到的274个关键词节点和由其组成的486条连线进行适当聚类(tf * idf结果显示分12类),绘制出可视化Timeline知识图谱(图 2)。由图 2分析可得,土壤中抗生素各研究主题间界限清晰(Q= 7 074,S=0.724 7),但关键词的整体网络结构(density= 0.013)较为松散,从而导致了各种小聚类的存在;土壤中抗生素研究的不同主题持续期各不相同,如规模最大的#0聚类自20世纪90年代之前便开始活跃,现在仍有研究持续进行;但也有一些研究主题持续较短,例如#12,这或许是由于在研究过程中找到了更深入本质的主题;并且图 2中显示的各研究主题间的颜色差异并不明显,表明1941—2018年并未出现时间跨度较大的主题研究阶段。第一个高频关键词于1991年出现(图中关键词所处的位置表示该研究首次出现的时间),直到90年代末开始涌现较多的高频关键词和交叉研究,可以得知土壤中抗生素的研究在近20年才被高度重视,结合关键词(图 2)及发文量(图 1)状况,可将该研究划分为三个阶段。
|
图 2 土壤中抗生素研究时间轴知识图谱 Figure 2 Timeline knowledge mapping of antibiotics in soil research |
(1)起步探索期(1941—1989年)。每年发表文章的数量基本无起伏(≤5篇·a-1),共发文63篇,占总发文量的4.65%,且基本无高频关键词的出现,可称作是土壤中抗生素研究的早期阶段。通过文献研读发现,该阶段的研究主要集中在土壤中新型抗生素的发现与分离。1940年,Waksman等[7]在筛选具有抗生素特性的土壤微生物时,首先发现了放线菌素,这是国内外学者对于土壤抗生素环境效应的初次系统探讨;紧接着,链丝菌素[10]、链霉素[11]以及新霉素[12]等20多种抗生素也相继被发现;随后,诸多学者分别在土壤微生物的研究中分离出了杆菌素[13]、氯霉素[14]、多肽类抗生素[15]、四环素[16]等多种新型抗生素。对分离的抗菌药物的化学性质[17]及其在医疗中实际应用的不断深入研究之后,仍然很少关注环境中可以分离出具有此类特殊功能化合物的原因[18],研究主题逐渐转向抗生素对土壤环境[19-20]、土壤微生物菌群[21-22]的影响分析;到此阶段后期,一些学者开始进行土壤中抗生素的检测条件探索[23-24],并认识到抗生素存在耐药性[25]。虽然此阶段的关键词并未集中到可以在知识图谱中呈现,但其对后续研究具有很强的奠基性,可以看出该研究领域的广泛范围。
(2)平稳发展期(1990—2002年)。相关文章的发表数量开始在一个区间内呈波动性上升趋势,即使在波谷期也仍有产出,保持研究的持续推进,共发文85篇,占总发文量的6.27%。经过早期的基础探索后,这段时间开始有多个重点研究主题出现,土壤中抗生素的研究逐渐趋向多元化,不仅继续鉴定新型抗生素生产者(土壤微生物)的生物学特性,也以痕量分析为基础探索土壤环境中多种抗生素的生态效应。抗生素在土壤环境中的转归[26]、吸附/解吸-降解等反应[27-29]以及环境风险评估参数和模型的计算与构建[30-31]等研究主题,在这一阶段得到了极大的发展,主要也得益于美国食品和药品管理局对环境中药品的风险过程评估和欧盟兽药的环境风险评估的政策推进。分析农业土壤及其施用的有机肥中的抗生素状况[32-33],成为了探求污染源、污染治理举措等细化研究的转折点。土壤中抗生素及其衍生物的检测方法[34]、抗生素的耐药性分析[35]在这一阶段也得到了持续关注,但抗性基因的研究尚未细化、深入。虽然此阶段涉及的聚类主题并不多(图 2),但各聚类间连线逐渐密集、关联变多,表明此阶段研究具有较强的影响力。
(3)快速增长期(2003—2018年)。每年发表的相关文章数量急速增加,共发文1 207篇,占总发文量的89.08%。这一阶段文献数量增长、研究主题增多、关联性增强,研究者尝试绘制抗生素在土壤环境中的“生命周期全图”[3, 36],并重视抗生素及其抗性基因所带来的世界性环境问题[37-38]。从主要研究内容来看,土壤中抗生素的“生命周期全图”研究集中在抗生素污染原因、影响因素及其迁移机理,分析了不同土地利用和土壤条件下的抗生素来源[39]、归趋特性[40],以及吸附[41]、迁移和降解[3]等过程中pH、阳离子和有机质等因素的作用,也对降低土壤中抗生素的含量、风险的关键措施[42-43]进行了深入探索,为解决土壤中多种抗生素所带来的毒理学挑战提供了基础。土壤中的抗生素抗性基因存在已久[6, 44],但借助分子生物学手段[45]开展其组成、积累[46]、迁移及影响因素[47-48]及其与土壤菌群结构和功能相互影响[49-50]的扩展研究,使得抗生素耐药性研究更加细化,取得极大的成效。本阶段的研究不仅注重科学技术的深入探索,也致力于解决抗生素滥用导致的一系列生态效应[51]和对人群健康的影响[52]等现实问题。
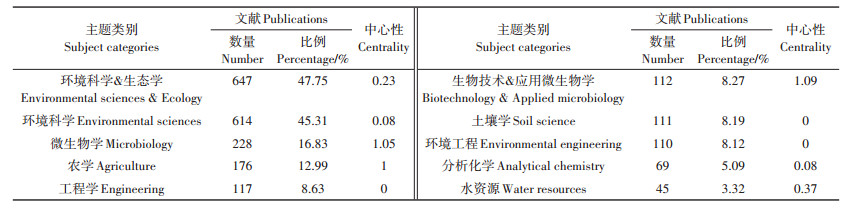
2.2 主要研究力量分析 2.2.1 发文主题类别分布对土壤中抗生素研究文献的主题类别进行总结(表 1),可知该研究方向受到多学科、多领域的广泛关注,环境科学&生态学是最大的刊出类别(647篇),占总刊出文章数的47.75%,随后是环境科学(614篇)占总数的45.31%,还涉及微生物学、农学等类别。生物技术&应用微生物学及微生物学的中心性较强;分别为1.09和1.05,表明它们在研究类别中的重要作用[53],主要是由于微生物学领域的关键技术是土壤中抗生素发现、分离及分析的重要保障[54]。但整体来看,土壤中抗生素的研究分布学科之间的交叉性较低[55],跨学科交叉研究可能会成为学者们今后研究的重要内容与主要突破点。
|
|
表 1 土壤中抗生素研究文献的主要类别 Table 1 The main subject categories of published articles of antibiotics in soil |
土壤中抗生素的研究作者共现图谱(图 3)可以反映该研究领域的核心作者、合作强度及其互引关系,可为评价研究学者的学术影响力提供参考[9, 55]。统计显示共有4 679名学者进行了土壤中抗生素的研究;借助于CiteSpace的作者共现分析工具,以TOP 50为阈值筛选出406名学者,整体合作关系结构较为松散(density=0.008 5),但在不同时期形成了以ThieleBruhn S,Smalla K,Wellington E M H,Groeneweg J,Zhu Y G,Ying G G等为中心的研究团队(图 3a);其中,Heuer H,Thiele-Bruhn S,Boxall A B A,Zhu Y G, Hamscher G,Sarmah A K和Hu X G的文章单篇引用量较高,且Thiele-Bruhn S,Boxall A B A,Kumar K等学者之间形成了较为密切的互引关系(图 3b)。

|
图 3 研究作者共现知识图谱 Figure 3 Co-occurrence knowledge mapping of research authors |
依据普莱斯定律确定核心作者,计算公式为M= 0.749(Nmax)1/2(其中Nmax为统计时间段内最高产作者的发文数量,M为核心作者的最低发文数量)。经统计,德国Thiele-Bruhn S教授是较早进行土壤中抗生素研究并且发文量最多(26篇)的学者,发文4篇以上(M=3.82)的学者可以认为是该研究领域的核心作者,且各核心作者合作团队成员之间联系紧密(Q= 0.961,S=0.727 6,图 3a)。
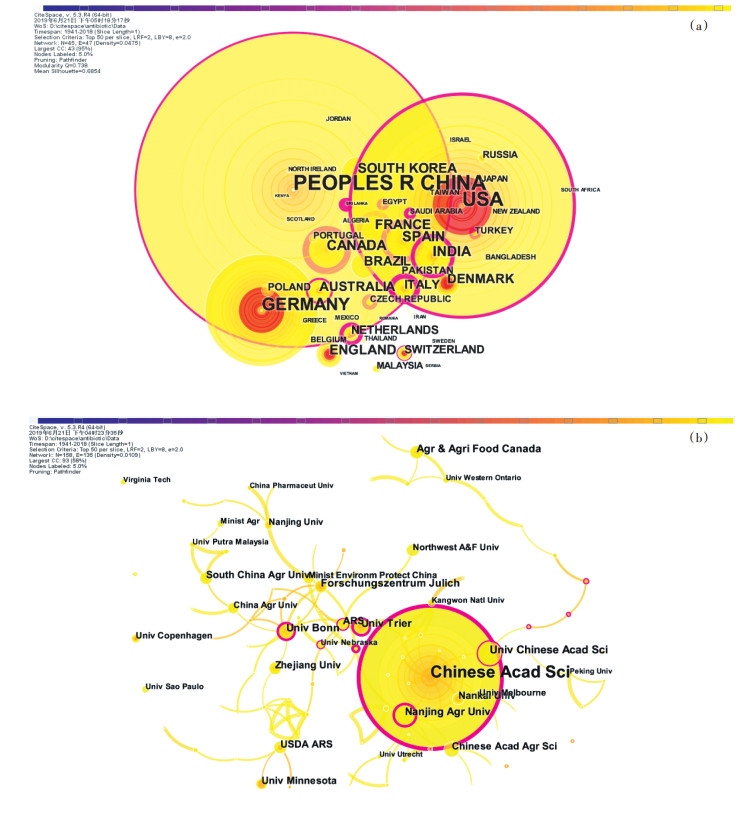
2.2.3 研究机构共现分析由图 4a可以看出,论文从属国家主要以中国、美国为中心形成聚类群,其中,中国的发文量最高(以节点大小标示),这可能归因于中国是一个农业大国,且农业拥有重要的战略地位;美国和德国在土壤抗生素研究中综合实力最强,英国、瑞士和丹麦均发布了影响力较强的研究成果(以红色圈进行标注);同时,加拿大的研究以最强的中介中心性(1.12)显示了其在研究关系网络中的重要作用[53],主要是由于加拿大研究者对环境中抗生素及抗性基因的全面认知及其预测模型研究中不可取代的重要贡献[56-57]。最大的发文从属机构聚类群(图 4b)是中国科学院(Chinese Acad Sci),其次以美国农业部农业工程应用技术研究所(USDA-ARS)、南京农业大学(Nanjing Agr Univ)、特里尔大学(Univ Trier)、波恩大学(Univ Bonn)、中国农业科学院(Chinese Acad Agr Sci)和浙江大学(Zhejiang Univ)等单位为交叉机构的合作成果较多。合并从属机构后,中国科学院、美国农业部、德国亥姆霍兹联合会、中国农业科学院、法国国家科研中心和南京农业大学的中介中心性较强,得益于这些研究机构对土壤中抗生素长期且系统的科学研究[48, 52]。
|
图 4 土壤中抗生素研究机构共现知识图谱 Figure 4 Co-occurrence knowledge mapping of antibiotics in soil research affiliations |
关键词是对文章主题的高度概括,研究领域内广受关注的核心问题探索会形成高频次、高中心性的文章关键词,可以借此来确定土壤中抗生素研究领域的热点[55]。
根据多诺霍提出的公式来划分高、低频词,公式为T=[-1+(1+8I)1/2]/2(T为高频词出现的最低次数,I为关键词的个数),得到土壤中抗生素研究领域的T为22.91,因此出现23次及以上的关键词是该领域的高频关键词,分别为soil(339次)、antibiotics(277次)、veterinary(264次)、adsorption(247次)等56个,高频关键词占总数的20.44%,可以看出土壤中抗生素研究的广泛性;高中介中心性的节点关键词为contaminant(0.37)、tylosin(0.26)、antibiotic(0.19)、diversity(0.18)、degradation(0.17)、sulfonamide antimicrobial agent(0.17)、chlortetracycline(0.15)、community(0.15)、bacteria(0.14)、clay(0.14)、waste water(0.13)、tetracycline(0.11)、antibiotic resistance(0.11)、aquatic environment(0.10)、biodegradation(0.10)。
综合高频次、高中心性关键词分布,可以基本确定土壤中抗生素的研究热点为:
(1)土壤中抗生素的种类及残留水平:固相萃取、微波辅助萃取、液相/气相色谱法、光谱法、层析法、串联质谱法以及液质联用等方法的不断改进,为在痕量水平上定性、定量及快速确定土壤中的抗生素提供了保障;
(2)土壤中抗生素的环境行为:对不同土地利用类型、不同性质的土壤(黏土、沙土等)中抗生素来源进行详细分析,并着重探索农业土壤中抗生素的吸附/解吸、迁移、降解(光解、水解和生物降解等)、转化等行为的反应过程及影响因素;
(3)土壤中抗生素的生态毒性及抗性基因:抗生素“假连续性”和抗性基因“可传递性”导致的一系列生态效应,也被众多学者和国际机构(组织)高度关注,侧重于生态风险评估方法的探索和抗性基因与土壤菌群的相互影响。
另外,CiteSpace的突显词(burst)探测算法可以探测在某一时间段内被引频次或共现频次突显度增加的节点(突显值的大小表现了其研究方向的重要性),以此来预测领域内的研究方向[55]。经探测得到36个突显词,其中出现最早的关键词为bacteria(8.046,1995年)、microorganism(4.626,1996年)、tylosin(8.216,2000年)和oxytetracycline(5.824,2000年),持续时间最长的关键词为microorganism(4.626,1996— 2008年)和tylosin(8.216,2000—2011年),突显值最大的关键词为antibiotic resistance gene(21.647)、abundance(11.531)和solid phase extraction(10.149),分析可得,研究热点主要出现在快速增长期(2003— 2018年),起步探索期(1941—1989年)并未出现突显词,平稳发展期(1990—2002年)虽然只出现4个突显词,但其与后期的研究热点衔接,可以看出该阶段研究的极大影响作用。土壤中抗生素的后续研究也会持续聚焦在痕量分析方法和抗生素抗性基因等主题。另外,对21世纪初兴起并持续保持热度的非突显关键词进行分析,可以发现区域尺度上土壤中抗生素残留的空间分析还处于发展阶段[58-61]。因此,借助GIS等空间分析软件对土壤中抗生素在面源上的空间分布进行研究,可有效实现对土壤抗生素污染的快速监测,控制大面积土壤抗生素生态危害,具有广泛的应用潜力。
3 结论本研究借助CiteSpace软件对土壤中抗生素研究领域的文献进行了计量学和可视化分析,梳理了其研究现状与发展历程,归纳了研究的核心与热点前沿方向,从整体上描绘了研究的总体样貌。
(1)土壤中抗生素的研究主要经历了起步探索期(1941—1989年)、平稳发展期(1990—2002年)和快速增长期(2003—2018年)三个阶段;研究方向由最初对土壤中新型抗生素的发现与分离逐渐转向多元化探索,不仅继续鉴定新型抗生素生产者(土壤微生物)的生物学特性,也以痕量分析为基础研究土壤环境中多种抗生素的生态效应,并尝试绘制抗生素在土壤环境中的“生命周期全图”,以更好地解决抗生素及其抗性基因所带来的世界性环境问题。
(2)土壤中抗生素的研究受到多学科、多领域的广泛关注,且跨学科的合作及知识的交叉融合使其相关的新兴领域和主题不断涌现,将成为后续研究的主要突破点;当前已形成联系紧密的核心作者群,且中国科学院发文量最多,而美国和德国的研究成果影响力最大。
(3)当前土壤中抗生素的研究主要侧重于种类及残留水平分析、环境行为模拟、生态毒性及抗性基因影响与控制等主题,但相对于水环境中的抗生素研究而言,土壤中抗生素及抗性基因的生态风险的关注度与研究力量尚显薄弱。
(4)土壤中抗生素的来源复杂,且影响其降解、迁移等行为的因素较多,应针对性地研究准确、高效的定量分析方法;另外,应综合考虑大区域尺度上的快速监测、风险评估及其与空间分析方法的结合,快速直观地呈现土壤中抗生素的空间分布特征;并完善土壤中抗生素及抗性基因的生态风险评估系统,探索土壤中抗生素高风险区域的治理与修复路径,以期为抗生素的合理使用与生态环境风险预估提供保障,并切实满足解决现实环境问题的实际需求。
| [1] |
Wassenaar T M. Use of antimicrobial agents in veterinary medicine and implications for human health[J]. Critical Reviews in Microbiology, 2004, 31(3): 155-169. |
| [2] |
Tasho R P, Cho J Y. Veterinary antibiotics in animal waste, its distribution in soil and uptake by plants:A review[J]. Science of the Total Environment, 2016, 563/564: 366-376. DOI:10.1016/j.scitotenv.2016.04.140 |
| [3] |
Chee-Sanford J C, Mackie R I, Koike S, et al. Fate and transport of antibiotic residues and antibiotic resistance genes following land application of manure waste[J]. Journal of Environmental Quality, 2009, 38(3): 1086-1108. DOI:10.2134/jeq2008.0128 |
| [4] |
Gavalchin J, Katz S E. The persistence of fecal-borne antibiotics in soil[J]. Journal of AOAC International, 1994, 77(2): 481-485. DOI:10.1093/jaoac/77.2.481 |
| [5] |
Vaclavik E, Halling-Sorensen B, Ingerslev F. Evaluation of manometric respiration tests to assess the effects of veterinary antibiotics in soil[J]. Chemosphere, 2004, 56(7): 667-676. DOI:10.1016/j.chemosphere.2004.02.018 |
| [6] |
Allen H K, Donato J, Wang H H, et al. Call of the wild:Antibiotic resistance genes in natural environments[J]. Nature Reviews Microbiology, 2010, 8(4): 251-259. DOI:10.1038/nrmicro2312 |
| [7] |
Waksman S A, Woodruff H B. Actinomyces antibiotics, a new soil organism antagonistic to pathogenic and non-pathogenic bacteria[J]. Journal of Bacteriology, 1941, 42(2): 231-249. DOI:10.1128/JB.42.2.231-249.1941 |
| [8] |
曹文杰, 赵瑞莹. 国际农业面源污染研究演进与前沿——基于CiteSpace的量化分析[J]. 干旱区资源与环境, 2019, 33(7): 1-9. CAO Wen-jie, ZHAO Rui-ying. Evolution and frontiers of international agricultural diffused pollution research:Quantitative analysis based on CiteSpace[J]. Journal of Arid Land Resources and Environment, 2019, 33(7): 1-9. |
| [9] |
安显金, 李维. 基于CNKI的我国生物炭研究趋势文献计量学分析[J]. 农业资源与环境学报, 2018, 35(6): 483-491. AN Xian-jin, LI Wei. Bibliometric analysis on research trend of biochar in China based on CNKI[J]. Journal of Agricultural Resources and Environment, 2018, 35(6): 483-491. |
| [10] |
Waksman S A, Woodruff H B. Streptothricin, a new selective bacteriostatic and bactericidal agent active against gram-negative bacteria[J]. Proceedings of the Society for Experimental Biology and Medicine, 1942, 49(2): 207-210. |
| [11] |
Schatz A, Bugie E, Waksman S A. Streptomycin, a substance exhibiting antibiotic activity against gram positive and gram-negative bacteria[J]. Proceedings of the Society for Experimental Biology and Medicine, 1944, 55(1): 66-69. |
| [12] |
Waksman S A, Lechevalier H A. Neomycin, a new antibiotic active against streptomycin-resistant bacteria, including tuberculosis organisms[J]. Science, 1949, 109(2830): 305-307. DOI:10.1126/science.109.2830.305 |
| [13] |
Foster J W, Woodruff H B. Bacillin, a new antibiotic substance from a soil isolate of Bacillus subtilis[J]. Journal of Bacteriology, 1946, 51(3): 363-369. DOI:10.1128/JB.51.3.363-369.1946 |
| [14] |
Ehrlich J, Bartz Q R, Smith R M, et al. Chloromycetin, a new antibiotic from a soil actinomycete[J]. Science, 1947, 106(2757): 417. DOI:10.1126/science.106.2757.417 |
| [15] |
Junowiczkocholaty R, Kocholaty W, Kelner A. Sulfactin, a new antibiotic substance produced by a soil actinomyces[J]. Journal of Biological Chemistry, 1947, 168(2): 765-769. |
| [16] |
Conover L H. Tetracycline: US 2699054[P]. 1953-10-09.
|
| [17] |
Pramer D. The persistence and biological effects of antibiotics in soil[J]. Applied Microbiology, 1958, 6(3): 221-224. DOI:10.1128/AEM.6.3.221-224.1958 |
| [18] |
Thaysen A C. Antibiotics in the soil[J]. Nature, 1950, 166(4211): 93-94. DOI:10.1038/166093a0 |
| [19] |
Nissen T V. Effects of antibiotics on carbon dioxide production in soil[J]. Nature, 1954, 174(4422): 226-227. DOI:10.1038/174226a0 |
| [20] |
Warman P R, Thomas R L. Chlortetracycline in soil amended with poultry manure[J]. Canadian Journal of Soil Science, 1981, 61(1): 161-163. DOI:10.4141/cjss81-017 |
| [21] |
Tsao P H, Thieleke D W. Stimulation of bacteria and actinomycetes by polyene antibiotic pimaricin in soil dilution plates[J]. Canadian Journal of Microbiology, 1966, 12(6): 1091-1094. DOI:10.1139/m66-148 |
| [22] |
Vaartaja O, Agnihotri V P. Interaction of nutrients and 4 antifungal antibiotics in their effects on pythium species in vitro and in soil[J]. Plant and Soil, 1969, 30(1): 49-61. DOI:10.1007/BF01885260 |
| [23] |
Witkamp M, Starkey R L. Some factors affecting determination of antibiotics in soil[J]. Soil Science, 1973, 115(5): 376-379. DOI:10.1097/00010694-197305000-00007 |
| [24] |
Katz J M, Katz S E. Microbial assay systems for determining antibiotic residues in soils[J]. Journal of the Association of Official Analytical Chemists, 1983, 66(3): 640-645. |
| [25] |
Fredrickson J K, Bezdicek D F, Brockman F J, et al. Enumeration of Tn5 mutant bacteria in soil by using a most-probable-number DNA hybridization procedure and antibiotic-resistance[J]. Applied and Environmental Microbiology, 1988, 54(2): 446-453. |
| [26] |
Halling-Sorensen B, Nielsen S N, Lanzky P F, et al. Occurrence, fate and effects of pharmaceutical substances in the environment:A review[J]. Chemosphere, 1998, 36(2): 357-394. DOI:10.1016/S0045-6535(97)00354-8 |
| [27] |
Bloom R A, Matheson J C. Environmental assessment of avermectins by the US Food and Drug Administration[J]. Veterinary Parasitology, 1993, 48(1/2/3/4): 218-294. |
| [28] |
Tolls J. Sorption of veterinary pharmaceuticals in soils:A review[J]. Environmental Science & Technology, 2001, 35(17): 3397-3406. |
| [29] |
Boxall A B A, Blackwell P, Cavallo R, et al. The sorption and transport of a sulphonamide antibiotic in soil systems[J]. Toxicology Letters, 2002, 131(1/2): 19-28. |
| [30] |
Spaepen K R I, VanLeemput L J J, Wislocki P G, et al. A uniform procedure to estimate the predicted environmental concentration of the residues of veterinary medicines in soil[J]. Environmental Toxicology and Chemistry, 1997, 16(9): 1977-1982. DOI:10.1002/etc.5620160930 |
| [31] |
Loke M L, Tjornelund J, Halling-Sorensen B. Determination of the distribution coefficient(logKd)of oxytetracycline, tylosin A, olaquindox and metronidazole in manure[J]. Chemosphere, 2002, 48(3): 351-361. DOI:10.1016/S0045-6535(02)00078-4 |
| [32] |
Loke M L, Ingerslev F, Halling-Sorensen B, et al. Stability of tylosin A in manure containing test systems determined by high performance liquid chromatography[J]. Chemosphere, 2000, 40(7): 759-765. DOI:10.1016/S0045-6535(99)00450-6 |
| [33] |
Haller M Y, Muller S R, McArdell C S, et al. Quantification of veterinary antibiotics(sulfonamides and trimethoprim)in animal manure by liquid chromatography-mass spectrometry[J]. Journal of Chromatography A, 2002, 952(1/2): 111-120. |
| [34] |
Hamscher G, Sczesny S, Hoper H, et al. Determination of persistent tetracycline residues in soil fertilized with liquid manure by high-performance liquid chromatography with electrospray ionization tandem mass spectrometry[J]. Analytical Chemistry, 2002, 74(7): 1509-1518. DOI:10.1021/ac015588m |
| [35] |
Halling-Sorensen B, Sengelov G, Tjornelund J. Toxicity of tetracyclines and tetracycline degradation products to environmentally relevant bacteria, including selected tetracycline-resistant bacteria[J]. Archives of Environmental Contamination and Toxicology, 2002, 42(3): 263-271. DOI:10.1007/s00244-001-0017-2 |
| [36] |
Sarmah A K, Meyer M T, Boxall A B A. A global perspective on the use, sales, exposure pathways, occurrence, fate and effects of veterinary antibiotics(VAs)in the environment[J]. Chemosphere, 2006, 65(5): 725-759. DOI:10.1016/j.chemosphere.2006.03.026 |
| [37] |
Forsberg K J, Reyes A, Bin W, et al. The shared antibiotic resistome of soil bacteria and human pathogens[J]. Science, 2012, 337(6098): 1107-1111. DOI:10.1126/science.1220761 |
| [38] |
Berendonk T U, Manaia C M, Merlin C, et al. Tackling antibiotic resistance:The environmental framework[J]. Nature Reviews Microbiology, 2015, 13(5): 310-317. DOI:10.1038/nrmicro3439 |
| [39] |
Biel-Maeso M, Corada-Fernandez C, Lara-Martin P A. Monitoring the occurrence of pharmaceuticals in soils irrigated with reclaimed wastewater[J]. Environmental Pollution, 2018, 235: 312-321. DOI:10.1016/j.envpol.2017.12.085 |
| [40] |
Kay P, Blackwell P A, Boxall A B A. Fate of veterinary antibiotics in a macroporous tile drained clay soil[J]. Environmental Toxicology and Chemistry, 2004, 23(5): 1136-1144. DOI:10.1897/03-374 |
| [41] |
Sassman S A, Lee L S. Sorption of three tetracyclines by several soils:Assessing the role of pH and cation exchange[J]. Environmental Science & Technology, 2005, 39(19): 7452-7459. |
| [42] |
Chen Q L, An X L, Zhu Y G, et al. Application of struvite alters the antibiotic resistome in soil, rhizosphere, and phyllosphere[J]. Environmental Science & Technology, 2017, 51(14): 8149-8157. |
| [43] |
Gou M, Hu H W, Zhang Y J, et al. Aerobic composting reduces antibiotic resistance genes in cattle manure and the resistome dissemination in agricultural soils[J]. Science of the Total Environment, 2018, 612: 1300-1310. DOI:10.1016/j.scitotenv.2017.09.028 |
| [44] |
D'Costa V M, King C E, Kalan L, et al. Antibiotic resistance is ancient[J]. Nature, 2011, 477(7365): 457-461. DOI:10.1038/nature10388 |
| [45] |
Su J Q, Wei B, Xu C Y, et al. Functional metagenomic characterization of antibiotic resistance genes in agricultural soils from China[J]. Environment International, 2014, 65: 9-15. DOI:10.1016/j.envint.2013.12.010 |
| [46] |
Zhu Y G, Johnson T A, Su J Q, et al. Diverse and abundant antibiotic resistance genes in Chinese swine farms[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(9): 3435-3440. DOI:10.1073/pnas.1222743110 |
| [47] |
Pu C, Liu H, Ding G, et al. Impact of direct application of biogas slurry and residue in fields:In situ analysis of antibiotic resistance genes from pig manure to fields[J]. Journal of Hazardous Materials, 2018, 344: 441-449. DOI:10.1016/j.jhazmat.2017.10.031 |
| [48] |
Zhu Y G, Gillings M, Simonet P, et al. Microbial mass movements[J]. Science, 2017, 357(6356): 1099-1100. DOI:10.1126/science.aao3007 |
| [49] |
Jechalke S, Focks A, Rosendahl I, et al. Structural and functional response of the soil bacterial community to application of manure from difloxacin-treated pigs[J]. FEMS Microbiology Ecology, 2014, 87(1): 78-88. |
| [50] |
Forsberg K J, Patel S, Gibson M K, et al. Bacterial phylogeny structures soil resistomes across habitats[J]. Nature, 2014, 509(7502): 612-616. DOI:10.1038/nature13377 |
| [51] |
Grenni P, Ancona V, Caracciolo A B. Ecological effects of antibiotics on natural ecosystems:A review[J]. Microchemical Journal, 2018, 136: 25-39. DOI:10.1016/j.microc.2017.02.006 |
| [52] |
Steffan J J, Brevik E C, Burgess L C, et al. The effect of soil on human health:An overview[J]. European Journal of Soil Science, 2018, 69(1): 159-171. |
| [53] |
陈超美, 陈悦, 侯剑华, 等. CiteSpaceⅡ:科学文献中新趋势与新动态的识别与可视化[J]. 情报学报, 2009, 28(3): 401-421. CHEN Chao-mei, CHEN Yue, HOU Jian-hua, et al. CiteSpace Ⅱ:Detecting and visualizing emerging trends and transient patterns in scientific literature[J]. Journal of the China Society for Scientific and Technical Information, 2009, 28(3): 401-421. |
| [54] |
Conover L H. Discovering tetracycline[J]. Research Management, 1984, 27(5): 17-22. |
| [55] |
陈悦, 陈超美, 刘则渊, 等. CiteSpace知识图谱的方法论功能[J]. 科学学研究, 2015, 33(2): 242-253. CHEN Yue, CHEN Chao-mei, LIU Ze-yuan, et al. The methodology function of CiteSpace mapping knowledge domains[J]. Studies in Science of Science, 2015, 33(2): 242-253. |
| [56] |
Boxall A B A, Rudd M A, Brooks B W, et al. Pharmaceuticals and personal care products in the environment:What are the big questions?[J]. Environmental Health Perspectives, 2012, 120(9): 1221-1229. DOI:10.1289/ehp.1104477 |
| [57] |
Larsson D G J, Andremont A, Bengtsson-Palme J, et al. Critical knowledge gaps and research needs related to the environmental dimensions of antibiotic resistance[J]. Environment International, 2018, 117: 132-138. DOI:10.1016/j.envint.2018.04.041 |
| [58] |
Xiang Q, Chen Q L, Zhu D, et al. Spatial and temporal distribution of antibiotic resistomes in a peri-urban area is associated significantly with anthropogenic activities[J]. Environmental Pollution, 2018, 235: 525-533. DOI:10.1016/j.envpol.2017.12.119 |
| [59] |
Shi W J, Yue T X, Du Z P, et al. Surface modeling of soil antibiotics[J]. Science of the Total Environment, 2016, 543: 609-619. DOI:10.1016/j.scitotenv.2015.11.077 |
| [60] |
Xie Y F, Li X W, Wang J F, et al. Spatial estimation of antibiotic residues in surface soils in a typical intensive vegetable cultivation area in China[J]. Science of the Total Environment, 2012, 430: 126-131. DOI:10.1016/j.scitotenv.2012.04.071 |
| [61] |
Sun J T, Zeng Q T, Tsang D C W, et al. Antibiotics in the agricultural soils from the Yangtze River Delta, China[J]. Chemosphere, 2017, 189: 301-308. DOI:10.1016/j.chemosphere.2017.09.040 |
 2020, Vol. 37
2020, Vol. 37