2. 农业农村部环境保护科研监测所, 天津 300191
2. Agro-Environmental Protection Institute, Ministry of Agriculture and Rural Affairs, Tianjin 300191, China
表观遗传学是基于非基因序列改变所致的基因表达水平的变化[1-3],表观遗传修饰手段主要包括DNA甲基化、组蛋白共价修饰、染色质重塑、基因印记和RNA干扰等五种[4],其中DNA甲基化是最重要的表观遗传修饰方式之一,也是目前机制研究最为透彻的表观遗传过程[5-7]。DNA甲基化是指在不改变DNA双螺旋序列的基础上,在DNA甲基转移酶的催化下将甲基转移到DNA分子中胞嘧啶残基上的过程。DNA甲基化存在器官、组织和发育时段的时空特异性[8],即不同器官、同一器官不同发育阶段之间的DNA甲基化模式和水平不同。在对拟南芥的子叶、叶片和花进行甲基化敏感扩增多态性(Methyla⁃ tion sensitive amplified polymorphism,MSAP)分析后发现,被检测到的甲基化模式存在着明显的器官特异性[9]。在拟南芥中,成熟叶片DNA甲基化水平比幼苗高20%,种子DNA甲基化水平又高于成熟叶片,但种子在发芽时发生去甲基化,甲基化水平又下降[10]。植物DNA甲基化模式的转换在维持植物正常生长发育过程中至关重要[11-12],在植物基因表达、基因组防御以及系统发育中起着重要的调节作用[13]。
黄顶菊(Flaveria bidentis)是2001年在天津市和河北省衡水湖先后被发现的一种重要外来入侵杂草,后扩散到河南、山东等多省[14-17]。黄顶菊凭借自身对环境极强的适应能力在新环境中比其他外地植物甚至是本地植物更具有资源竞争优势,除了具有较强的生物学特性,基因组DNA甲基化的改变很有可能是调控入侵植物生境适应能力的重要机制之一,然而目前对黄顶菊基因组DNA甲基化的分布模式、变异情况等还缺乏全面的认识。如前所述,植物不同组织和不同发育阶段的DNA甲基化模式和水平是不同的,植物的不同器官、同一器官的不同发育阶段在DNA甲基化模式上均可能存在差异。在研究黄顶菊表观遗传学与其入侵性的关系之前,应该充分考虑其DNA甲基化的时空变化是否存在特异性。因此,本实验建立一个合理的MSAP体系来研究黄顶菊不同器官和同一器官不同发育阶段的表观遗传变化特征,为黄顶菊环境适应性获得的表观遗传机制提供理论基础,并从表观遗传学角度为科学采样策略提供思路。
1 材料和方法 1.1 试验设计和样品采集本试验在农业农村部环境保护科研监测所网室(39°05′N,117°08′E)内进行。2015年4月底,于天津静海团泊洼水库附近采集长势均一且生长旺盛的黄顶菊植株,移栽至网室花盆(规格为28 cm×18 cm×24 cm)中,每盆装土10 kg,每3日浇水一次,土壤由原土和草炭混合而成,每7日转盆一次以消除环境干扰,移苗60日后采集实验材料。样品采集方法:
(1) 黄顶菊不同器官组织的采集。随机筛选20株长势一致的植株,每株采集从上至下第5对无病虫害且完全展开的叶片,用于黄顶菊叶组织的DNA甲基化检测;采集第5节和第6节之间去节间的茎用于黄顶菊茎组织的DNA甲基化检测;把整株黄顶菊植株连根拔起,抖落土壤并清洗干净,采集第一级侧根用于黄顶菊根组织DNA甲基化的检测。
(2) 黄顶菊不同发育阶段器官组织的采集。选取同一植株从上至下第2对生长健康且完全展开的叶片作为黄顶菊嫩叶,第5对叶片作为黄顶菊老叶来研究同一器官不同发育阶段的表观遗传变化特征。以上样品采集后均迅速用锡纸包裹并放入液氮中速冻,随后转入-70 ℃冰箱贮存,用于后续DNA甲基化的检测。
1.2 甲基化MSAP体系建立 1.2.1 DNA提取为满足MSAP体系对DNA高质量的要求,参照王鹤潼等[18]CTAB法并对其进行改进,提取黄顶菊不同组织器官的基因组DNA。
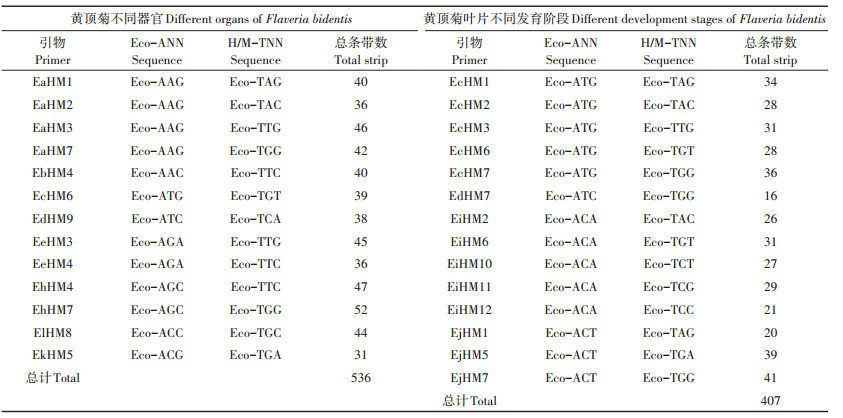
1.2.2 MSAP体系建立与优化将提取的基因组DNA依次进行酶切连接、预扩增、选择性扩增3个PCR体系的扩增反应[19-20]。PCR体系所用的接头和引物序列见表 1。
|
|
表 1 接头和引物序列信息 Table 1 Sequence information of adaptor and primers |
制备5% PAGE凝胶,待其完全凝固后经过电泳、银染、显色过程,晾干后扫描并进行条带计数和分析。
1.3 数据统计与分析采用Quantity One软件将5%变性PAGE凝胶100~500 bp范围的扩增条带进行标记并转化成0/1矩阵。用POP Gene软件分析引物的多样性指数。用SPSS 17软件和Origin 9.1软件分析并绘制黄顶菊各器官、各发育阶段主成分及甲基化状态图,更加直观地反映黄顶菊各器官及各发育阶段表观遗传多样性的变化。
2 结果与分析 2.1 黄顶菊DNA提取和PAGE凝胶电泳提取高纯度DNA是本实验成功的关键,MSAP技术中样本基因组DNA必须满足OD260/OD280为1.7~ 1.9,本试验中黄顶菊根、茎和叶组织基因组DNA的OD260/OD280分别为1.78、1.82和1.88,不同发育阶段老叶和嫩叶组织基因组DNA的OD260/OD280分别为1.84和1.86,满足实验要求。进一步用1%琼脂糖凝胶电泳检测,电泳图中基因组DNA(图 1)条带亮、无杂质、无降解,可用于后续MSAP反应体系。

|
图 1 黄顶菊不同器官(A)及不同发育阶段(B)基因组DNA提取 Figure 1 Genomic DNA extraction of the Flaveria bidentis in different organs(A)and different development stages(B) |
经MSAP体系扩增获得的混合产物通过5%变性PAGE凝胶电泳分离,部分引物的甲基化条带检测结果如图 2所示,基因组扩增条带清晰且均匀,可用于扩增条带的统计与分析。本研究选用13对引物通过MSAP体系对黄顶菊不同器官进行甲基化检测,条带统计结果(表 2)显示共扩增出536条条带,平均每对引物扩增41条,其中EhHM7引物扩增获得的条带数最多,为52条,而EkHM5引物扩增的条带数最少,仅获得31条条带。通过MSAP体系选用14对引物对黄顶菊不同发育阶段组织进行甲基化检测(表 2),共扩增出407条MSAP条带,平均每对引物扩增29条,其中EjHM7引物扩增获得的条带数最多,而EdHM7引物获得的条带数最少,分别为41条和16条。

|
图 2 黄顶菊不同器官(A)及不同发育阶段(B)DNA MSAP检测结果 Figure 2 Results of genomic DNA MSAP detection in different organs(A)and different developmental stages(B)of Flaveria bidentis |
|
|
表 2 黄顶菊不同器官及不同发育阶段引物组合的序列信息及扩增条带数 Table 2 Sequence information and amplification number of primer combinations in different organs and different development stages of Flaveria bidentis |
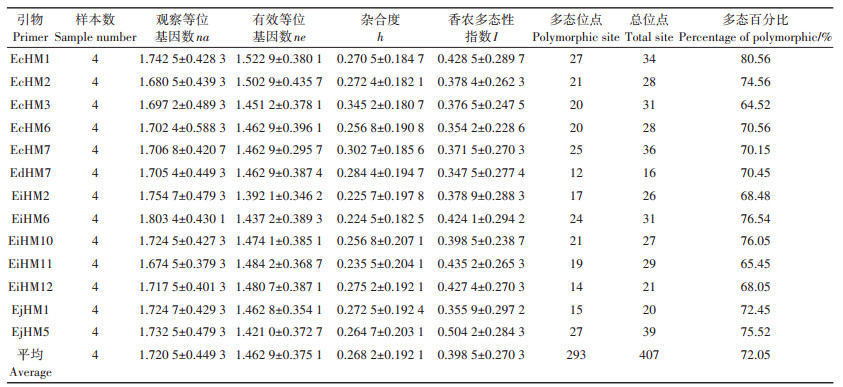
如表 3所示,黄顶菊不同器官MSAP体系筛选的引物组合中观察等位基因数(na)平均值为1.842 6,有效等位基因数(ne)为1.623 1,杂合度(h)为0.345 0,香农多态性指数(I)为0.500 4。13对引物共扩增出536个位点,其中多态性位点为451个,多态性百分比为84.26%。全部引物组合的等位基因数、有效等位基因数、杂合度、香农多态性指数均高于平均值,引物多样性指数综合指标偏高,其中EhHM7对表观遗传多样性贡献率最大,多态性百分比为92.45%;EcHM6的贡献率最小,多态性百分比为78.84%。如表 4所示,黄顶菊叶器官不同发育阶段MSAP体系筛选的引物组合中观察等位基因数平均值为1.720 5,有效等位基因数为1.462 9,杂合度为0.268 2,香农多态性指数为0.398 5。选用的14对引物的总扩增位点数为407,其中多态性位点为293个,多态性百分比为72.05%。EcHM1对表观遗传多样性贡献率最大,多态性百分比为80.56%;EcHM3的贡献率最小,多态性百分比为64.52%。
|
|
表 3 黄顶菊不同器官引物的遗传多样性分析 Table 3 Primer genetic diversity analysis of different organs of Flaveria bidentis |
|
|
表 4 黄顶菊叶片不同发育阶段引物的遗传多样性分析 Table 4 Primer genetic diversity analysis of Flaveria bidentis leaves at different development stages |
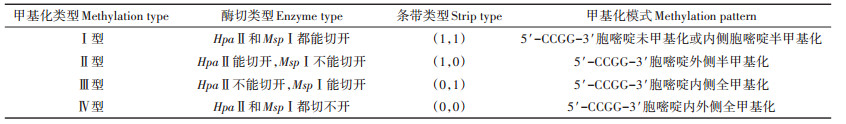
HpaⅡ和MspⅠ两种内切酶均可以识别真核生物中的甲基化位点,即5′-CCGG -3′位点,其中5′-CCGG -3′位点能被内切酶切开记作1,不能被内切酶切开记作0,据此可以将MSAP甲基化模式分为4种类型(表 5)。Ⅰ类型甲基化状态为未甲基化,记作(1,1);Ⅱ类型甲基化状态为半甲基化,记作(1,0);Ⅲ类型甲基化状态为全甲基化,记作(0,1);Ⅳ类型甲基化状态信息不确定,记作(0,0)。
|
|
表 5 MSAP甲基化模式类型 Table 5 MSAP methylation pattern types |
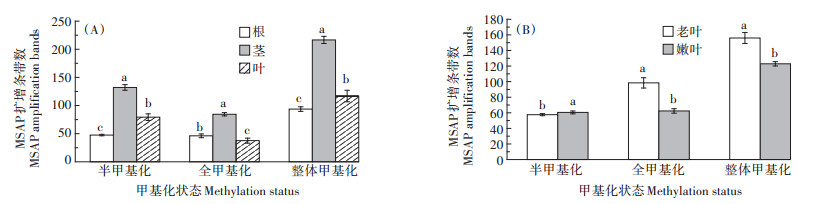
由图 3可知,黄顶菊不同器官(根、茎和叶)间以及同一器官不同发育阶段(老叶和嫩叶)组织间的甲基化水平均表现出显著性差异(P < 0.05)。各甲基化变异类型中,茎组织的甲基化发生率最高。其中,在发生半甲基化变异类型的各组织中,茎组织的甲基化发生率为30.28%,分别较叶组织和根组织高出12.63、19.74个百分点;全甲基化变异类型中,茎组织甲基化发生率为19.37%,分别较根组织和叶组织高出9.11、11.00个百分点;整体甲基化变异类型中,茎组织甲基化发生率为49.66%,分别较叶组织和根组织高出23.64、28.86个百分点;老叶组织的全甲基化和整体甲基化的发生率分别为33.29%和52.77%,分别较嫩叶组织高出13.59、13.96个百分点。
|
同种甲基化状态下不同字母表示差异显著(P < 0.05) Different letters for the same methylation status indicate significant differences(P < 0.05) 图 3 黄顶菊不同器官(A)和不同发育阶段(B)的甲基化状态 Figure 3 Methylation status of Flaveria bidentis in different organs(A)and different development stages(B) |
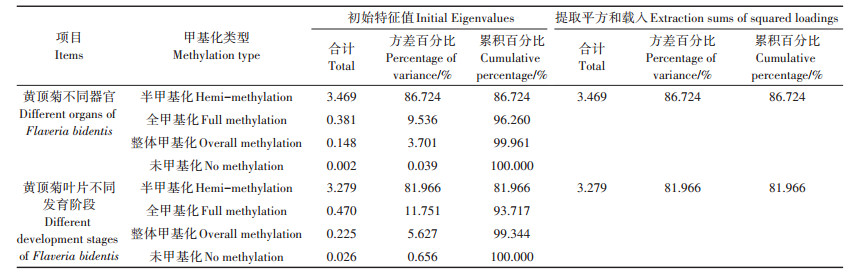
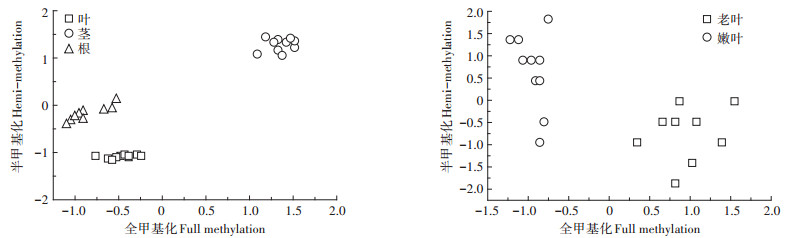
通过对黄顶菊各器官及同一器官叶片不同发育阶段的总方差分析(表 6),可知其半甲基化、全甲基化、整体甲基化和未甲基化公因子方差分别为0.892、0.894、0.702、0.981和0.669、0.909、0.739、0.961,数值接近则说明变量中大部分信息可用于结果分析,真实性较高。不同器官的主成分分析有且只有一个特征值3.469(>1),可知半甲基化模式在甲基化水平中起主导作用。从图 4可知,黄顶菊不同器官根、茎、叶各组织10个个体分别聚集在一起,形成了3个较为集中的区域,每个区域中个体间距离较近,说明其亲缘关系和甲基化变异类型都相近。根、茎和叶各个区域之间分布存在一定距离,可知不同器官间的甲基化模式有差异。黄顶菊相同器官个体间相互比较,可知叶片个体分布较根、茎的分布更为密集,意味着黄顶菊根、茎的个体间差异大于叶片个体间差异。黄顶菊叶片不同的发育阶段主成分分析只有一个特征值3.279 (>1),嫩叶和老叶聚集区域显著,但两者分布距离较远,且各自聚集程度并不紧密,说明存在明显的个体间差异,这种个体间差异不可忽视。因此在进行DNA甲基化相关研究时,为得出缜密且可靠的实验结果,必须制定科学的采样方案,将植物不同器官以及相同器官不同发育阶段之间的甲基化特异性考虑在内。
|
|
表 6 黄顶菊不同器官和不同发育阶段解释总方差 Table 6 Interpretation of total variance of different organs and leaves at different development stages of Flaveria bidentis |
|
图 4 黄顶菊不同器官和不同发育阶段主成分分析 Figure 4 Principal component analysis of different organs and different development stages of Flaveria bidentis |
黄顶菊是入侵我国华北地区最严重的杂草之一,为控制其蔓延发生,国内外学者对其入侵机制进行了广泛探究,但已有研究多集中在对不同环境条件下黄顶菊的种子萌发特性、形态、化感效应强弱及生理生化指标变化规律方面[21-25,16],很少从表观遗传学角度研究其生态适应性机理[26]。非生物胁迫下植物可以通过甲基化水平及模式的改变调节其基因表达,从而提高其适应性,DNA甲基化变异很可能是调控入侵植物新生境适应能力的重要途径之一,但黄顶菊各器官组织基因组DNA甲基化的相关研究报道还很少。DNA甲基化在植物体内随植物生长发育发生时空变化,包括同一植物的不同器官和同一器官的不同发育阶段[7]。本研究中,通过对甲基化类型条带的统计,发现黄顶菊不同组织根、茎、叶及同一器官不同发育阶段老叶和嫩叶之间的甲基化水平均表现出显著性差异,说明黄顶菊在生长发育过程中其甲基化状态是一个动态过程,不同器官和同一器官不同发育阶段甲基化存在时空特异性,可能是其适应环境的不同于传统遗传学的一种内在机制。这与Finnegan等[10]最早对拟南芥的研究结果一致,他们认为拟南芥成熟叶片的甲基化水平较嫩叶高出20%,但成熟叶片的甲基化水平又显著低于种子。
经过主成分分析发现,黄顶菊各组织甲基化水平及变异类型存在时空特异性,其不同器官之间亲缘关系较远,且根的个体间差异较大。黄顶菊老叶和嫩叶之间亲缘关系较远,且老叶和嫩叶个体间差异较为明显。施雯[27]对喜旱莲子草和刺花莲子草的叶和茎两种器官DNA甲基化的研究中同样发现,两种莲子草的DNA甲基化具有器官特异性,且两种莲子草的器官差异性均大于个体间差异。喜旱莲子草的不同发育阶段甲基化差异大于个体间差异,而刺花莲子草两者差异相当。陆光远等[28]研究发现油菜的不同器官胚根、下胚轴和子叶的甲基化水平依次呈现显著升高的趋势,且种子在萌发过程中甲基化模式也发生变化,也说明植物在生长发育过程中甲基化模式及水平是一个动态的过程。由此可见,研究植物的表观遗传变化特征需要考虑器官的特异性差异及同一器官个体间的差异性,这种差异不可忽视。
4 结论(1) 各甲基化变异类型中黄顶菊不同器官中茎组织的甲基化发生率最高,在全甲基化和整体甲基化模式中黄顶菊不同发育阶段甲基化发生率表现为老叶 > 嫩叶,黄顶菊不同器官(根、茎和叶)间以及同一器官不同发育阶段(老叶和嫩叶)的组织间均表现出显著的甲基化水平差异性,这可能是其适应新生境的表观遗传学机理之一。
(2) 黄顶菊根个体间、茎个体间、老叶与嫩叶个体间均存在较大的差异性,在采样过程中应充分考虑植物器官和生长发育阶段特异性。
| [1] |
Mirouze M, Paszkowski J. Epigenetic contribution to stress adaptation in plants[J]. Current Opinion in Plant Biology, 2011, 14: 1-8. DOI:10.1016/j.pbi.2010.11.003 |
| [2] |
Yan S, Rui H, Fu X, et al. Genome-wide analysis of DNA methylation in five tissues of Zhikong scallop, Chlamys farreri[J]. PLoS ONE, 2014, 9(1): e86232. DOI:10.1371/journal.pone.0086232 |
| [3] |
Richards C L, Schrey A W, Pigliucci M. Invasion of diverse habitats by few Japanese knotweed genotypes is correlated with epigenetic differentiation[J]. Ecology Letters, 2012, 15(9): 1016-1025. DOI:10.1111/j.1461-0248.2012.01824.x |
| [4] |
凡时财, 张学工. DNA甲基化的生物信息学研究进展[J]. 生物化学与生物物理进展, 2009, 36(2): 143-150. FAN Shi-cai, ZHANG Xue-gong. Progress of bioinformatics study in DNA methylation[J]. Prog Biochem Biophys, 2009, 36(2): 143-150. |
| [5] |
李新玲, 徐香玲. 植物DNA甲基化与表观遗传[J]. 中国农学通报, 2008, 24(1): 123-126. LIN Xin-ling, XU Xiang-ling. DNA methylation in plants and its epigenetic[J]. Chinese Agricultural Science Bulletin, 2008, 24(1): 123-126. |
| [6] |
李娜, 张旸, 解莉楠, 等. 植物DNA甲基化研究进展[J]. 植物生理学报, 2012, 48(11): 1027-1036. LI Na, ZHANG Yang, XIE Li-nan, et al. Research progress in DNA methylation in plants[J]. Plant Physiology Journal, 2012, 48(11): 1027-1036. |
| [7] |
唐晓梅, 王艳, 马东伟, 等. 干旱胁迫下高羊茅基因组甲基化分析[J]. 草业学报, 2015, 24(4): 164-173. TANG Xiao-mei, WANG Yan, MA Dong-wei, et al. Methylation analysis of tall fescue genome under drought stress[J]. Acta Prataculturae Sinica, 2015, 24(4): 164-173. |
| [8] |
Iwasaki M, Paszkowski J. Identification of genes preventing transgenerational transmission of stress-induced epigenetic states[J]. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(23): 8547-8552. DOI:10.1073/pnas.1402275111 |
| [9] |
Ruizgarcía L, Cervera M T, Martínezzapater J M. DNA methylation increases throughout Arabidopsis development[J]. Planta, 2005, 222(2): 301-306. DOI:10.1007/s00425-005-1524-6 |
| [10] |
Finnegan E J, Genger R K, Kovac K, et al. DNA methylation and the promotion of flowering by vernalization[J]. Proceedings of the National Academy of Sciences of the United States of America, 1998, 95(10): 5824-5829. DOI:10.1073/pnas.95.10.5824 |
| [11] |
Sahu P P, Pandey G, Sharma N, et al. Epigenetic mechanisms of plant stress responses and adaptation[J]. Plant Cell Reports, 2013, 32(8): 1151-1159. DOI:10.1007/s00299-013-1462-x |
| [12] |
Marconi G, Pace R, Traini A, et al. Use of MSAP markers to analyse the effects of salt stress on DNA methylation in rapeseed(Brassica napus var. oleifera)[J]. PLoS ONE, 2013, 8(9): e75597. DOI:10.1371/journal.pone.0075597 |
| [13] |
Feng W, Dong Z, He B, et al. Analysis method of epigenetic DNA methylation to dynamically investigate the functional activity of transcription factors in gene expression[J]. BMC Genomics, 2012, 13(1): 532. DOI:10.1186/1471-2164-13-532 |
| [14] |
李香菊, 王贵启, 张朝贤, 等. 外来植物黄顶菊的分布、特征特性及化学防除[J]. 杂草科学, 2006(4): 58-61. LI Xiang-ju, WANG Gui-qi, ZHANG Chao-xian, et al. The distribution of alien plants Flaveria bidentis, characteristics and chemical control[J]. Weed Science, 2006(4): 58-61. DOI:10.3969/j.issn.1003-935X.2006.04.024 |
| [15] |
郭成亮, 胡文多, 朱敏峰, 等. 有害杂草黄顶菊在河北衡水的入侵途径调查[J]. 植物检疫, 2007, 2(3): 187-188. GUO Cheng-liang, HU Wen-duo, ZHU Min-feng, et al. Harmful weeds Flaveria bidentis in Hebei Hengshui invading route survey[J]. Plant Quarantine, 2007, 2(3): 187-188. DOI:10.3969/j.issn.1005-2755.2007.03.021 |
| [16] |
芦站根, 崔兴国, 蒋文静. 衡水湖黄顶菊的入侵情况的初步调查研究[J]. 衡水学院学报, 2006, 8(1): 69-71. LU Zhan-gen, CUI Xing-guo, JIANG Wen-jing. The primary investigation and studies on the alien invasion of Flaveria Bidentis Kuntze in Hengshui Lake[J]. Hengshui College Journal, 2006, 8(1): 69-71. DOI:10.3969/j.issn.1673-2065.2006.01.026 |
| [17] |
赵晓红, 杨殿林, 王慧, 等. 黄顶菊入侵对不同地区土壤氮循环及微生物量的影响[J]. 草业学报, 2015, 24(2): 62-69. ZHAO Xiao-hong, YANG Dian-lin, WANG Hui, et al. Effects of the invasion of Flaveria bidentis on soil nitrogen cycling and microbial biomass in different regions[J]. Acta Pratacultura Sinica, 2015, 24(2): 62-69. |
| [18] |
王鹤潼, 何蕾, 宋杰, 等. 改进MSAP-RCR技术应用于Cd胁迫下拟南芥DNA甲基化分析[J]. 农业环境科学学报, 2015, 34(8): 1618-1624. WANG He-tong, HE Lei, SONG Jie, et al. Assay of DNA methylation in Arabidopsis under Cd stress using improved MSAP-PCR technique[J]. Journal of Agro-Environment Science, 2015, 34(8): 1618-1624. |
| [19] |
张燕, 陈波. DNA甲基化敏感扩增多态性技术及其在作物遗传研究中的应用[J]. 西昌学院学报, 2009, 12(7): 7-11. ZHANG Yan, CHEN Bo. DNA methylation-sensitive amplified polymorphism and its application in plant genetics[J]. Xichang College Journal, 2009, 12(7): 7-11. |
| [20] |
陈瑞娟, 何蕾, 孙梨宗, 等. 铜胁迫对拟南芥幼苗生长和基因组DNA甲基化的影响[J]. 生态学杂志, 2014, 19(4): 2650-2657. CHEN Rui-juan, HE Lei, SUN Li-zong, et al. Effects of copper stress on the growth and genomic DNA methylation of Arabidopsis thaliana seedlings[J]. Chinese Journal of Ecology, 2014, 19(4): 2650-2657. |
| [21] |
Huangfu C, Li H, Chen X, et al. Response of an invasive plant, Flaveria bidentis, to nitrogen addition:A test of form-preference uptake[J]. Biological Invasions, 2016, 18(11): 1-16. |
| [22] |
Tetu S G, Tanz S K, Vella N, et al. The Flaveria bidentis beta-carbonic anhydrase gene family encodes cytosolic and chloropliastic isoforms demonstrating distinct organ-specific expression patterns[J]. Plant Physiology, 2007, 144(3): 1316-1327. DOI:10.1104/pp.107.098152 |
| [23] |
乔建国, 张玉蕊, 康利芬. 黄顶菊物候和种子发芽特征研究[J]. 现代园林, 2008, 12: 84-86. QIAO Jian-guo, ZHANG Yu-rui, KANG Li-fen. Study on phenology and seed germination characteristics of Flaveria bidentis[J]. Modern Garden, 2008, 12: 84-86. |
| [24] |
任艳萍, 江莎, 古松, 等. 外来入侵植物黄顶菊研究进展[J]. 热带亚热带植物学报, 2008, 6(4): 390-396. REN Yan-ping, JIANG Sha, GU Song, et al. Research progress of invasive plants of Flaveria bidentis[J]. Journal of Tropical and Subtropical Botany, 2008, 6(4): 390-396. DOI:10.3969/j.issn.1005-3395.2008.04.019 |
| [25] |
樊翠芹, 王贵启, 李秉华, 等. 黄顶菊的生长繁殖特性[J]. 杂草科学, 2008(3): 37-39. FAN Cui-qin, WANG Gui-qi, LI Bing-hua, et al. The growth and reproduction characteristics of Flaveria bidentis[J]. Weed Science, 2008(3): 37-39. DOI:10.3969/j.issn.1003-935X.2008.03.010 |
| [26] |
全志星, 田佳源, 张思宇, 等. 不同入侵地区黄顶菊DNA表观遗传多样性变化特征[J]. 农业环境科学学报, 2017, 36(4): 625-634. QUAN Zhi-xing, TIAN Jia-yuan, ZHANG Si-yu, et al. The epigenetic diversity of DNA of Flaveria bidentis in different invasive areas[J]. Journal of Agro-Environment Science, 2017, 36(4): 625-634. |
| [27] |
施雯.植物DNA甲基化的器官特异性及其对生态遗传学采样策略的启示[D].昆明: 云南大学, 2012. SHI Wen. Organ specificity of plant DNA methylation and its implications for ecological genetic sampling strategies[D]. Kunming: Yunnan University, 2012. http://cdmd.cnki.com.cn/Article/CDMD-10673-1012419166.htm |
| [28] |
陆光远, 伍晓明, 陈碧云, 等. 油菜种子萌发过程中DNA甲基化的MSAP分析[J]. 科学通报, 2005, 50(24): 2750-2756. LU Guang-yuan, WU Xiao-ming, CHEN Bi-yun, et al. MSAP analysis of DNA methylation in rape seed germination[J]. Chinese Science Bulletin, 2005, 50(24): 2750-2756. DOI:10.3321/j.issn:0023-074X.2005.24.012 |
 2019, Vol. 36
2019, Vol. 36