文章信息
- 李吉祥, 吴文良, 郭岩彬
- LI Ji-xiang, WU Wen-liang, GUO Yan-bin
- 细菌生物合成纳米硒的研究进展
- Review on Nano SeleniumProduced by Bacteria
- 农业资源与环境学报, 2014, 31(6): 551-554
- Journal of Agricultural Resources and Environment, 2014, 31(6): 513-520
- http://dx.doi.org/10.13254/j.jare.2014.0245
-
文章历史
- 收稿日期:2014-09-25
硒(Se)是人和动物体内必需的一种微量元素,与人体内一系列的抗氧化功能、甲状腺激素代谢和免疫系统等密不可分[1]。但硒在高浓度会产生毒害作用,污染环境。自然界中的硒循环与细菌的活动紧密相关[2],许多细菌可将氧化态硒转化为毒性较小、活性较高的红色纳米硒(red nano-elemental selenium,NSe)[3],这种生物纳米硒应用潜力巨大。
近年来对硒的研究主要集中在硒在土壤中的分布转化、在植物中的吸收转化、筛选耐硒菌等,而在生物纳米硒及细菌生成纳米硒机制方面研究极少。本文主要综述了纳米硒、细菌合成纳米硒机理及影响因素等方面的研究进展,并分析了未来该研究的关注点,为今后研究细菌与纳米硒的相互关系及生物纳米硒的广泛应用提供参考。 1 纳米硒的特性及应用
纳米硒具有很好的光电学和半导体特性,已广泛应用于太阳能电池、整流器、传感器等光电元件[4]。纳米硒颗粒与C=O,COO和C-N组蛋白结合时会表现出重要的生物活性[5],可用于生物、化学、医药等领域,比如可以生产硒-维生素、食品添加剂、新型抗生素衣料,以及在防治癌症方面的应用[6, 7]等。
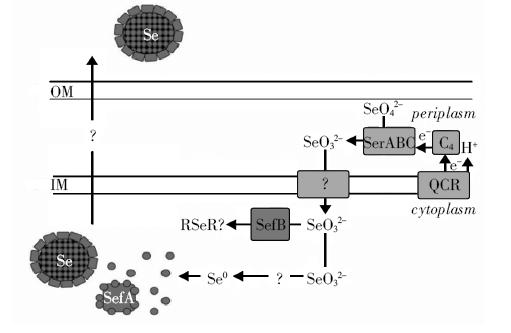
纳米硒大体可分为工业纳米硒和生物纳米硒两类。工业纳米硒可通过化学试剂(如肼、硫代硫酸钠、维生素C等)氧化还原反应、电化学技术、结晶技术等生产,但这些方法受高温、高压、催化剂等技术限制[8],同时生产的纳米硒稳定性差,大小不均一,生物活性极低[9]。通过细菌转化形成的生物纳米硒大小较为均一,为高电子密度[10]。同时发现SefA(selenium factor A)调控蛋白能够控制红色元素硒的聚合,将蛋白包裹在纳米硒表面,使得纳米硒性质更稳定,活性更高(图 1)[11]。Dobias等[8]在大肠杆菌中分离出4种特异性蛋白(ACEA-异柠檬酸裂解酶,IDH-异柠檬酸脱氢酶[NADP],OMPC-外膜蛋白,ADHP-乙醇脱氢酶)结合在纳米硒(Red elemental selenium)上面,其中ADHP蛋白可调控纳米硒使其大小、形态均一,但这4种蛋白在纳米硒形成中的具体机理还不清楚。无毒高效的生物纳米硒在植物上的应用也表现出良好的生物学效应。Domokos等[12]在2012年研究纳米硒对烟草植物的生物效应影响时发现,生物纳米硒在265~530 μmol·L-1浓度范围内能够刺激烟草器官的发育形成,并能够增加烟草根系的生长,而硒酸盐在任何浓度下都不具有上述生物学效应。
研究发现,许多变形菌门和厚壁菌门的细菌具有将硒酸盐或亚硒酸盐还原为单质纳米硒的能力,其中既有化能异养菌也有光合细菌,有好氧菌也有厌氧菌[13],如大肠杆菌(Escherichia coli)、枯草芽孢杆菌(Bacillus subtilis)、蜡样芽孢杆菌(Bacillus cereus)、地衣芽孢杆菌(Bacillus licheniformis)、荧光假单胞菌(Pseudomonas fluorescens)等。但是不同的生长条件会对细菌合成纳米硒产生影响。 2.1 碳、氮源对微生物还原硒的影响
对枯草芽孢杆菌和荧光假单胞菌还原亚硒酸盐的研究时发现纳米单质硒的生成效果因培养基中碳源的不同而不同,其中以葡萄糖为碳源时的还原效果最为明显[14]。同时,部分细菌对亚硒酸盐的还原效率及还原量也会随着碳源量的增加而上升[15]。这可能是由于碳源是细菌生长的物质基础,不同的碳源及含量会影响细菌的生长繁殖,继而会影响细菌还原硒酸盐的能力。
而在不同的氮源作为电子受体的情况下,微生物还原硒的速率和还原量也会受到影响。Losi等[16]分别以NO3-,SeO32-,NO3-+SeO32-作为电子受体时发现NO3-在一定的含量条件下会明显抑制阴沟肠杆菌(Enterobacter cloacae)还原硒的速率和还原量,这可能是由于NO3- 在与SeO32-同时存在的情况下会竞争电子或降低了硒酸盐/亚硒酸盐还原酶的活性[17]。但是,Sabaty等[18]研究表明在NO3-含量相对少的情况下反而会促进硒的还原,则是因为硒的还原特性还会受到周质亚硝酸还原酶的作用。Zhang等[15]研究在硒污水中影响肠杆菌(Enterobacter taylorae)还原硒的因素实验中也证明了上述结论。 2.2 氧气对微生物还原硒的影响
因为细菌还原硒盐是一个氧化还原过程,所以氧气含量会影响细菌生成纳米硒的过程。Bebien[19]在研究亚硒酸根离子对球形红细菌的生长和蛋白生成量的影响时发现,其在好氧条件下对亚硒酸盐的转化率为25%,而在厌氧条件下几乎全部被转化。而且,在好氧条件下,球形红细菌中的SOD酶、含巯基蛋白和硫氧还蛋白的量会迅速增加,但在厌氧条件下这些酶或蛋白的含量较低。所以,球形红细菌在厌氧条件与好氧条件下其产生红色纳米硒的机理可能不同。 2.3 温度、pH值及CO2含量对微生物还原硒的影响
温度、pH值也会影响微生物还原硒的效率。Bao等[17]在不同的温度和pH值条件下检测梭菌芽孢杆菌(Clostridium)对硒酸盐和亚硒酸盐的转化效率时发现,这种菌在30 ℃和pH=7的条件下转化效率最高。而Lortie等[20]发现施氏假单胞菌(Pseudomonas stutzeri)在30 ℃左右和pH=8的弱碱性环境中转化效率最高。
同时,CO2的含量在一定程度上会增加梭菌芽孢杆菌(Clostridium)对亚硒酸盐的转化效率[17]。一方面可能是CO2会增加这种菌的生长量或者是增强了菌内氢化酶I的活性;另一方面,可能是CO2会诱导与膜结合的b-type细胞色素的表达,有利于周质空间中的电子传导,从而加速了亚硒酸盐的还原。而且,CO2也可能通过这种方式增加硒酸盐的还原速率。 3 细菌还原产生纳米硒的机制
微生物在硒的转化过程中起着重要的作用,目前,越来越多的细菌被发现可将硒还原生成纳米硒。但是,不同的细菌由于其所含酶、结构等的不同,其还原机理可能不同。Kuroda等[21]将其还原过程分为两个独立的过程:硒酸盐还原为亚硒酸盐和亚硒酸盐还原为纳米单质硒。其中研究硒酸盐被还原为亚硒酸盐的机理已较为透彻,硒酸盐的还原借助异化硫还原途径[22],通过可溶性周质硒酸盐还原酶系统(Ser ABC)[23]和钼离子结合蛋白参与完成,其中,细胞色素C4负责将电子从对苯二酚-细胞色素C还原酶(QCR)传递给SerABC系统,反应在周质空间内完成(图 1)。Yee 等[24]在阴沟肠杆菌(Enterobacter cloacae) SLD1a-1还原硒酸盐产生纳米硒的研究中发现,厌氧条件下局性转录调控基因FNR(延胡索酸盐硝酸还原酶)可调控硒酸盐还原酶活性,并且其FNR基因缺失突变体不能合成纳米硒,而互补菌体则恢复了合成纳米硒的能力。但亚硒酸盐被还原为单质硒的机制仍处于探索阶段(图 1)。
许多研究表明多种酶参与催化亚硒酸盐还原为纳米单质硒,如硝酸盐还原酶[25]、氢化酶I [26]、砷酸盐还原酶[27],甚至在一些细菌中亚硒酸盐的还原不需要酶的参与[28]。
Li等[29]最新研究发现沙雷菌(Shewanella oneidensis)MR-1在无氧呼吸中的富马酸还原酶(FccA)对亚硒酸盐的还原影响最大,其主要依靠中心细胞色素C(CymA)的调控,而其他酶如硝酸还原酶、MTR半胱氨酸簇蛋白酶等在还原过程中几乎不起作用。NADPH/NADH依赖还原酶也可能在还原过程中作为电子载体对亚硒酸盐的还原起作用[10]。
但一般认为,细菌通过酶催化还原亚硒酸盐的过程是在细胞质内进行的(图 1)。其可能的两种反应过程如下:
(1)硫化物介导的亚硒酸盐还原反应。1996年,Nelson等[30]通过体外试验发现,酒色着色菌(Chromatium vinosum)可将还原态硫(S2-)氧化成零价硫颗粒储存在胞内,当外界小分子如H2Se进入胞内后便与单质硫颗粒发生氧化还原反应生成一种硒-硫颗粒(S0+Se2-→Se0+S2-)。2003年,Hockin等[31]发现脱硫微菌(Desulfomicrobiumnorvegicum)可通过硫酸盐还原途径将硫酸盐还原为硫化物(S2-),并释放到胞外,当环境中存在亚硒酸根(SeO32-)时,S2-便与SeO32-反应,生成硒-硫颗粒,黏附于细胞表面(SeO32-+2S2-+6H+→Se0+2S0+ 3H2O)。
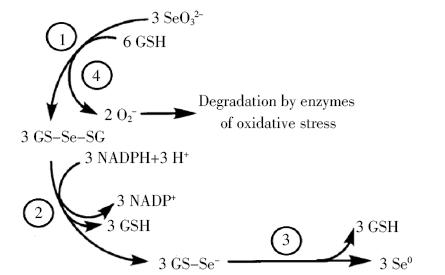
(2)谷胱甘肽GSH介导的反应。亚硒酸盐进入细菌后会与谷胱甘肽反应生成selenodiglutathione(GS-Se-SG)(图 2-①),GS-Se-SG在谷胱甘肽还原酶的作用下生成GS-Se-(图 2-②),而GS-Se-不稳定,会继续与氢离子反应产生较稳定的红色单质硒(图 2-③),其亚硒酸盐在被谷胱甘肽还原为单质硒的过程中还会产生超氧根离子(O2-)(图 2-④),O2-正是亚硒酸盐产生生物毒性的原因[32]。至今,这种方式被广泛认为是生物还原亚硒酸盐的主要模式。
总之,对于不同的微生物细菌可能会有不同的合成纳米硒的方式,还需进一步的研究找到其反应机制的普遍性规律。 4 总结
由于细菌具有生长迅速、繁殖快、代谢能力强、适应性强等特点,且利用微生物转化硒不受季节和气候的影响,生产周期短,无污染,故利用细菌生产纳米硒将成为未来的新热点,其应用也将越来越广泛,前景广阔。
目前,对于微生物细菌还原产生纳米硒的机理仍不清楚,许多相关性问题也未回答。未来仍需要在以下几个方面进行进一步的研究。一是确定纳米硒在细菌体内生成的位置,到底是在胞质、胞外还是胞膜上,或者是在这些位置内都有反应;二是找到控制纳米硒形成的关键性基因和酶,以及与其相关的调控系统,甚至有些蛋白也会参与其中,有许多调控重金属代谢的因子可能也会调控硒的代谢,是否具有这种普遍性还需研究去揭示;三是阐明纳米硒的运输方式及硒进入细菌的途径。如果纳米硒的形成是在胞质内,那么亚硒酸盐如何进入细胞,纳米硒又如何转出细胞的,还需要进一步研究。
| [1] | Stranges S, Marshall J R, Trevisan M, et al. Effects of selenium supplementation on cardiovascular disease incidence and mortality: secondary analyses in a randomized clinical trial[J]. American Journal of Epidemiology, 2006, 163(8): 694-699. |
| [2] | Haudin C S, Fardeau M L, Amenc L, et al. Responses of anaerobic bacteria to soil amendment with selenite[J]. Soil Biology and Biochemistry, 2007, 39(9): 2408-2413. |
| [3] | Zhang J, Wang H, Bao Y, et al. Nano red elemental selenium has no size effect in the induction of seleno-enzymes in both cultured cells and mice[J]. Life Sciences, 2004, 75(2): 237-244. |
| [4] | Zhang J, Zhang S Y, Xu J J, et al. A new method for the synthesis of selenium nanoparticles and the application to construction of H2O2 biosensor[J]. Chinese Chemical Letters, 2004, 15(11): 1345-1348. |
| [5] | Zhang S Y, Zhang J, Wang H Y, et al. Synthesis of selenium nanoparticles in the presence of polysaccharides[J]. Materials Letters, 2004, 58(21): 2590-2594. |
| [6] | Wang H L, Zhang J S, Yu H Q. Elemental selenium at nano size possesses lower toxicity without compromising the fundamental effect on selenoenzymes: comparison with selenomethionine in mice[J]. Free Radical Biology and Medicine, 2007, 42(10): 1524-1533. |
| [7] | Ramamurthy C H, Sampath K S, Arunkumar P, et al. Green synthesis and characterization of selenium nanoparticles and its augmented cytotoxicity with doxorubicin on cancer cells[J]. Bioprocess and Biosystems Engineering, 2013, 36(8): 1131-1139. |
| [8] | Dobias J, Suvorova E I, Bernier-Latmani R. Role of proteins in controlling selenium nanoparticle size[J]. Nanotechnology, 2011, 22(19): 195605. |
| [9] | 黄 峙, 郑文杰, 李乐农, 等. 纳米元素硒的生物合成及生物活性[J]. 中国生物工程杂志, 2003(7): 76-79. HUANG Zhi, ZHENG Wen-jie, LI Le-nong, et al. Research progress on biosynthesis and bioactivity of nano red elemental selenium[J]. China Biotechnology, 2003(7): 76-79.(in Chinese) |
| [10] | Dhanjal S, Cameotra S S. Aerobic biogenesis of selenium nanospheres by Bacillus cereus isolated from coalmine soil[J]. Microbial Cell Factories, 2010, 9: 52. |
| [11] | Debieux C M, Dridge E J, Mueller C M, et al. A bacterial process for selenium nanosphere assembly[J]. Proceedings of the National Academy of Sciences, 2011, 108(33): 13480-13485. |
| [12] | Domokos-Szabolcsy E, Marton L, Sztrik A, et al. Accumulation of red elemental selenium nanoparticles and their biological effects in Nicotinia tabacum[J]. Plant Growth Regul, 2012, 68(3): 525-531. |
| [13] | 郑世学, 粟 静, 王 瑞, 等. 硒是双刃剑——谈微生物中的硒代谢[J]. 华中农业大学学报, 2013, 32(5): 1-8. ZHENG Shi-xue, SU Jing, WANG Rui, et al. Metabolism of selenium in microorganism[J]. Journal of Huazhong Agricultural University, 2013, 5(32): 1-8.(in Chinese) |
| [14] | 王东亮, 肖 敏, 钱 卫, 等. 细菌还原氧化态硒产生红色单质硒的研究进展[J]. 微生物学报, 2007(3): 554-557. WANG Dong-liang, XIAO Min, QIAN Wei, et al. Progress in research of the product of the red elemental selenium reduced from selenium oxyanions by bacteria[J]. Acta Microbiologica Sinica, 2007(3): 554-557.(in Chinese) |
| [15] | Zhang Y, Zahir Z A, Frankenberger W T. Factors affecting reduction of selenate to elemental selenium in agricultural drainage water by Enterobacter taylorae[J]. Journal of Agricultural and Food Chemistry, 2003, 51(24): 7073-7078. |
| [16] | Losi M E, Frankenberger W T. Reduction of selenium oxyanions by Enterobacter cloacae SLD1a-1: isolation and growth of the bacterium and its expulsion of selenium particles[J]. Applied and Environmental Microbiology, 1997, 63(8): 3079-3084. |
| [17] | Bao P, Huang H, Hu Z Y, et al. Impact of temperature, CO2 fixation and nitrate reduction on selenium reduction, by a paddy soil Clostridium strain[J]. Journal of Applied Microbiology, 2013, 114(3): 703-712. |
| [18] | Sabaty M, Avazeri C, Pignol D, et al. Characterization of the reduction of selenate and tellurite by nitrate reductases[J]. Applied and Environmental Microbiology, 2001, 67(11): 5122-5126. |
| [19] | Bebien M, Chauvin J P, Adriano J M, et al. Effect of selenite on growth and protein synthesis in the phototrophic bacterium Rhodobacter sph-aeroides[J]. Applied and Environmental Microbiology, 2001, 67(10): 4440-4447. |
| [20] | Lortie L, Gould W D, Rajan S, et al. Reduction of selenate and selenite to elemental selenium by a Pseudomonas stutzeri isolate[J]. Applied and Environmental Microbiology, 1992, 58(12): 4042-4044. |
| [21] | Kuroda M, Yamashita M, Miwa E, et al. Molecular cloning and characterization of the srdBCA operon, encoding the respiratory selenate reductase complex, from the selenate-reducing bacterium Bacillus selenatarsenatis SF-1[J]. Journal of Bacteriology, 2011, 193(9): 2141-2148. |
| [22] | Hockin S, Gadd G M. Removal of selenate from sulfate containing media by sulfate reducing bacterial biofilms[J]. Environmental Microbiology, 2006, 8(5): 816-826. |
| [23] | Schröder I, Rech S, Krafft T, et al. Purification and characterization of the selenate reductase from Thauera selenatis[J]. Journal of Biological Chemistry, 1997, 272(38): 23765-23768. |
| [24] | Yee N, Ma J, Dalia A, et al. Se (VI) reduction and the precipitation of Se (0) by the facultative bacterium Enterobacter cloacae SLD1a-1 are regulated by FNR[J]. Applied and Environmental Microbiology, 2007, 73(6): 1914-1920. |
| [25] | Avazéri C, Turner R J, Pommier J, et al. Tellurite reductase activity of nitrate reductase is responsible for the basal resistance of Escherichia coli to tellurite[J]. Microbiology, 1997, 143(4): 1181-1189. |
| [26] | Yanke L J, Bryant R D, Laishley E J. Hydrogenase I of Clostridium pasteurianum functions as a novel selenite reductase[J]. Anaerobe, 1995, 1(1): 61-67. |
| [27] | Afkar E, Lisak J, Saltikov C, et al. The respiratory arsenate reductase from Bacillus selenitireducens strain MLS10[J]. FEMS Microbiology Letters, 2003, 226(1): 107-112. |
| [28] | Tomei F A, Barton L L, Lemanski C L, et al. Reduction of selenate and selenite to elemental selenium by Wolinella succinogenes[J]. Canadian Journal of Microbiology, 1992, 38(12): 1328-1333. |
| [29] | Li D B, Cheng Y Y, Wu C, et al. Selenite reduction by Shewanella oneidensis MR-1 is mediated by fumarate reductase in periplasm[J]. Scientific Reports, 2014, DOI: 10. 1038/serpo 37 35 |
| [30] | Nelson D C, Casey W H, Sison J D, et al. Selenium uptake by sulfur-accumulating bacteria[J]. Geochimica et Cosmochimica Acta, 1996, 60(18): 3531-3539. |
| [31] | Hockin S L, Gadd G M. Linked redox precipitation of sulfur and selenium under anaerobic conditions by sulfate-reducing bacterial biofilms[J]. Applied and Environmental Microbiology, 2003, 69(12): 7063-7072. |
| [32] | Kessi J, Hanselmann K W. Similarities between the abiotic reduction of selenite with glutathione and the dissimilatory reaction mediated by Rhodospirillum rubrum and Escherichia coli[J]. Journal of Biological Chemistry, 2004, 279(49): 50662-50669. |
 2014, Vol. 31
2014, Vol. 31